


生物技术进展 ›› 2024, Vol. 14 ›› Issue (5): 712-723.DOI: 10.19586/j.2095-2341.2024.0112
• 进展评述 • 上一篇
王晶( ), 关海涛, 张晓磊, 王堡槐, 刘宝海(
), 关海涛, 张晓磊, 王堡槐, 刘宝海( ), 温洪涛(
), 温洪涛( )
)
收稿日期:2024-06-11
接受日期:2024-08-01
出版日期:2024-09-25
发布日期:2024-10-22
通讯作者:
刘宝海,温洪涛
作者简介:王晶E-mail: buyijingjing@163.com;
基金资助:
Jing WANG( ), Haitao GUAN, Xiaolei ZHANG, Baohuai WANG, Baohai LIU(
), Haitao GUAN, Xiaolei ZHANG, Baohuai WANG, Baohai LIU( ), Hongtao WEN(
), Hongtao WEN( )
)
Received:2024-06-11
Accepted:2024-08-01
Online:2024-09-25
Published:2024-10-22
Contact:
Baohai LIU,Hongtao WEN
摘要:
基因编辑技术是重要的生物育种技术之一。随着生物育种产业化的高速发展,农业基因编辑产品呈现快速增长态势,然而其标识溯源检测技术的研发速度难以匹配知识产权保护和安全监管的需求,严重制约了产业的发展,亟需研发与现行检测体系相适应的基因编辑产品检测技术与方法。从主要的基因编辑系统和技术优势、靶点编辑类型着手,分析了不同类型基因编辑产品检测技术的特征和优缺点,提出了检测技术创新、检测与评价技术融合、监管技术体系细化三方面建议,以期为农业基因编辑产品标识、溯源技术研究提供参考,为完善我国农业生物技术产品监管体系提供技术支撑。
中图分类号:
王晶, 关海涛, 张晓磊, 王堡槐, 刘宝海, 温洪涛. 农业基因编辑产品检测动态及发展趋势[J]. 生物技术进展, 2024, 14(5): 712-723.
Jing WANG, Haitao GUAN, Xiaolei ZHANG, Baohuai WANG, Baohai LIU, Hongtao WEN. Detection Dynamic and Development Tendency of Agricultural Gene Editing Products[J]. Current Biotechnology, 2024, 14(5): 712-723.
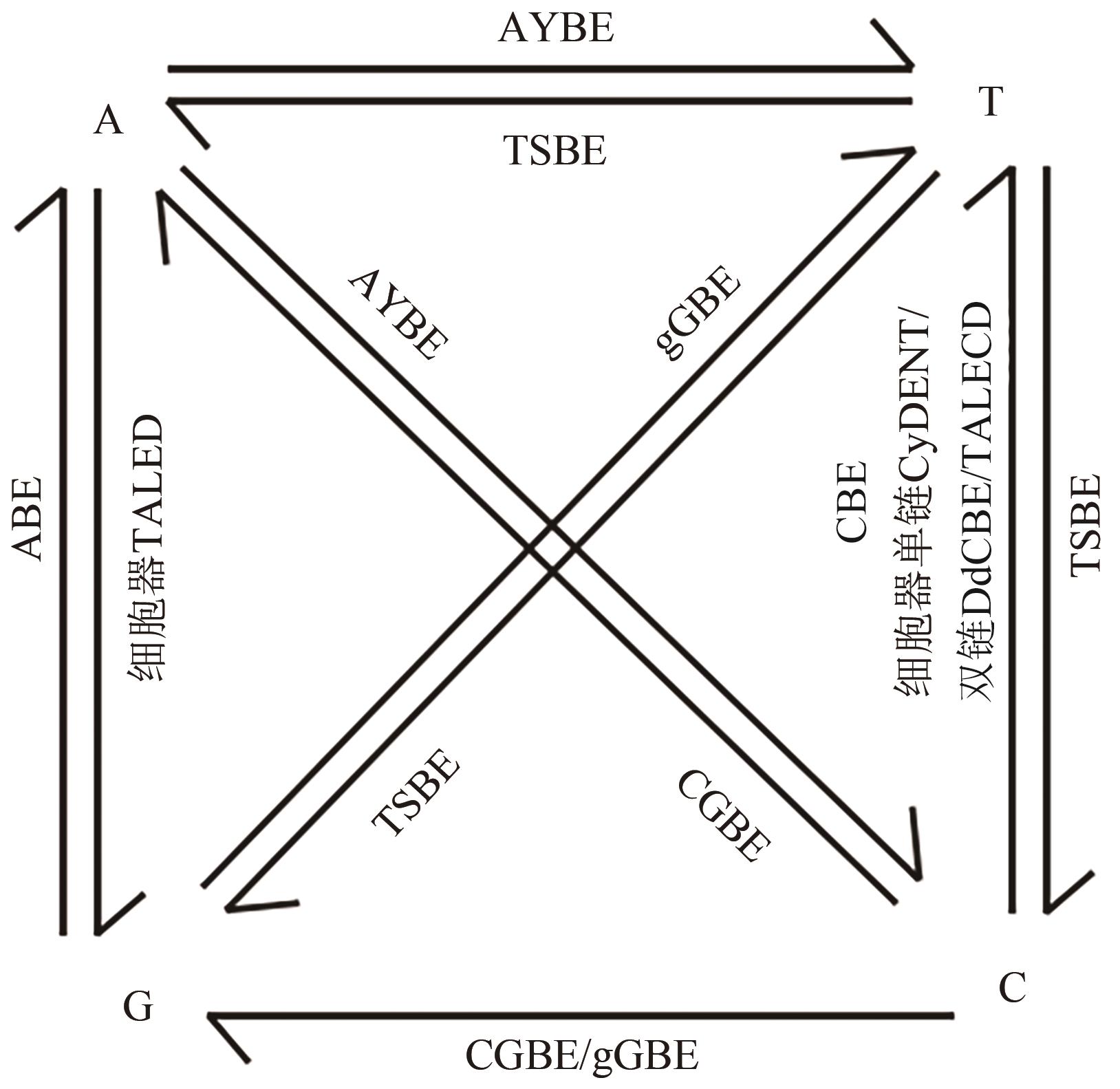
图1 主要单碱基编辑类型[12]注:ABE—腺嘌呤碱基编辑器(adenine base editor);AYBE—腺嘌呤颠换碱基编辑器(adenine transversion base editor);CBE—胞嘧啶碱基编辑器(cytosine base editor);CGBE—C→G碱基编辑器;gGBE—基于糖基化酶的鸟嘌呤碱基编辑器(glycosylase-based guanine base editor);TSBE—T→S(G/C)碱基编辑器。
Fig. 1 Main types of base editing
| 分类 | 方法 | 技术特征 | 优点 | 缺点 | 灵敏度/识别碱基 | 参考文献 |
|---|---|---|---|---|---|---|
| 基于普通PCR方法 | PCR/限制性核酸内切酶实验 | 编辑位点位于限制性酶切位点 | 简单、快速、经济,可检测纯合和杂合突变体 | 受限于原始序列 | - | [ |
| CAPS衍生的方法和indCAPS方法 | 编辑位点可以利用错配引物创制限制性酶切位点,利用网络工具indCAPS设计含indel的dCAPS引物 | 对于不同类型的indel具有更好的灵活性 | 需要设计特别的引物区分一致的indel等位 | - | [ | |
| T7核酸内切酶Ⅰ实验 | T7核酸内切酶降解野生型和突变型形成的错配的杂合双链 | 简单、快速、经济,能够检测杂合突变 | 灵敏度低,不能检测纯合突变 | - | [ | |
| 连接检测反应 | 使用高保真DNA连接酶将完全匹配的碱基对探针连接在两个合成寡核苷酸的缺口连接处时,等位基因特异性连接产物可以通过使用不同长度的引物和/或荧光标签修饰来唯一区分 | 低成本、精准、可靠,可以区分纯合与杂合基因型和多个等位基因 | 不能用于突变筛选 | 1 bp | [ | |
| 混合异源双链迁移率实验 | 基于有错配和无错配的DNA分子的差异迁移率,提供了一种简单方便的方法在测序确认之前辨别杂合生物体 | 可快速、有效地、筛选相关突变,简化纯合突变体分离鉴定过程 | 仅用于纯合突变体筛选 | - | [ | |
| 单链构象多态性 | 检测单链核酸,PCR扩增产物变性形成单链,非变性PAGE胶检测 | 可鉴定纯合突变 | 片段小于300 bp,不能区分编辑类型 | 10%,2 bp | [ | |
| 基于聚丙烯酰胺凝胶电泳的方法 | 同源双链DNA在天然PAGE中的迁移速度快于异源双链DNA | 简单、快速、经济,能够检测纯合和杂合突变 | 耗时长、低通量 | 0.5%,2 bp | [ | |
| 限制性片段长度多态性结合一代测序技术 | 取决于靶基因内是否产生限制性内切酶的识别位点 | 可测定突变类型 | 更费时、费力 | - | [ | |
| 扩增片段长度多态性结合一代测序技术 | 利用限制性内切酶水解基因组DNA,再使双链人工接头的酶切片段相连接,作为扩增反应的模板DNA,接头的互补链为引物进行预扩增,通过聚丙烯酰胺凝胶电泳分离检测 | 分辨率高、稳定性好、效率高 | 费用高,对DNA的纯度和内切酶的质量要求很高 | - | [ | |
| 临界温度退火PCR | PCR中临界退火温度抑制引物与模板的不匹配退火,抑制扩增子产生 | 简单、快速、经济,能够检测纯合突变 | 需要设计特异引物耗时费力 | 20%,1 bp | [ | |
| 特异性末端介导的聚合酶链式反应 | 设计产生带有特定末端部分的分子构建体,根据是否能够自折叠形成引物启动PCR扩增定性定量鉴定 | 能够定性定量检测单碱基突变,成本低、快速 | 引物设计复杂 | 30拷贝·反应-1 | [ | |
| 基于qPCR方法 | 实时荧光PCR和标记的实验 | 针对突变位点设计不同修饰类型探针,结合qPCR技术 | 简单、有效、灵敏 | 不能明确突变中核酸的变化 | <10%,1 bp | [ |
| 高分辨率熔解曲线分析技术 | 纯合DNA具有独特的退火温度,而突变的杂合DNA具有较低的熔化温度 | 快速有效检测SNP和indel类型的编辑 | 需要特殊的仪器,灵敏度受扩增产物长度影响 | 4.7%,2 bp | [ | |
| 基于聚合酶链式反应的高分辨片段分析方法 | 通过设计大小不同并用不同荧光团标记的PCR扩增子来对具有一个以上靶向基因的样品进行多重分析 | 灵敏度高,可检测到小至1 bp,能够区分多个突变的等位基因 | 大小相同的indel等位不能共同被洗脱和分辨 | 2%,1 bp | [ | |
| 简单等位基因辨别聚合酶链式反应 | 利用一种位点特异性反向引物,通用荧光探针和猝灭剂,以及热启动DNA聚合酶 | 快速、精准、低成本 | 需要提前优化PCR反应条件 | 0.4 ng·10 μL-1 | [ | |
| 基于数字PCR方法 | 数字PCR方法 | 将样品分割到许多单独的qPCR反应中,阴性结果用于生成样品中目标分子精确数量的绝对结果,无须参考标准品或内源性对照品 | 精准、低含量核酸加工食品、多倍体 | 成本高,需要特殊设备,不适用于大片段突变 | 1/100 000 | [ |
| 基于测序技术 | 全基因组测序 | 建库进行高通量测序 | 鉴定突变打靶、打靶类型和脱靶 | 成本高、耗时,可能存在内生菌或病原物序列信息的污染等 | - | [ |
| Sanger测序技术 | 第一代测序技术 | 准确鉴定突变位点 | 成本高、耗时、费力 | - | [ | |
| 基于其他技术 | CRISPR/Cas系统 | 利用CRISPR/Cas12特异性识别编辑位点和非特异性切割的功能 | 低成本、条件温和、操作简单、灵敏度高、能实现快速检测 | 可能存在脱靶效应导致假阳性或假阴性,需要PAM/PFS识别序列,难以标准化,需要扩增过程 | 12 拷贝·μL-1,1 bp | [ |
表1 基因编辑位点鉴定方法[32, 51]
Table 1 Identification method of gene editing site
| 分类 | 方法 | 技术特征 | 优点 | 缺点 | 灵敏度/识别碱基 | 参考文献 |
|---|---|---|---|---|---|---|
| 基于普通PCR方法 | PCR/限制性核酸内切酶实验 | 编辑位点位于限制性酶切位点 | 简单、快速、经济,可检测纯合和杂合突变体 | 受限于原始序列 | - | [ |
| CAPS衍生的方法和indCAPS方法 | 编辑位点可以利用错配引物创制限制性酶切位点,利用网络工具indCAPS设计含indel的dCAPS引物 | 对于不同类型的indel具有更好的灵活性 | 需要设计特别的引物区分一致的indel等位 | - | [ | |
| T7核酸内切酶Ⅰ实验 | T7核酸内切酶降解野生型和突变型形成的错配的杂合双链 | 简单、快速、经济,能够检测杂合突变 | 灵敏度低,不能检测纯合突变 | - | [ | |
| 连接检测反应 | 使用高保真DNA连接酶将完全匹配的碱基对探针连接在两个合成寡核苷酸的缺口连接处时,等位基因特异性连接产物可以通过使用不同长度的引物和/或荧光标签修饰来唯一区分 | 低成本、精准、可靠,可以区分纯合与杂合基因型和多个等位基因 | 不能用于突变筛选 | 1 bp | [ | |
| 混合异源双链迁移率实验 | 基于有错配和无错配的DNA分子的差异迁移率,提供了一种简单方便的方法在测序确认之前辨别杂合生物体 | 可快速、有效地、筛选相关突变,简化纯合突变体分离鉴定过程 | 仅用于纯合突变体筛选 | - | [ | |
| 单链构象多态性 | 检测单链核酸,PCR扩增产物变性形成单链,非变性PAGE胶检测 | 可鉴定纯合突变 | 片段小于300 bp,不能区分编辑类型 | 10%,2 bp | [ | |
| 基于聚丙烯酰胺凝胶电泳的方法 | 同源双链DNA在天然PAGE中的迁移速度快于异源双链DNA | 简单、快速、经济,能够检测纯合和杂合突变 | 耗时长、低通量 | 0.5%,2 bp | [ | |
| 限制性片段长度多态性结合一代测序技术 | 取决于靶基因内是否产生限制性内切酶的识别位点 | 可测定突变类型 | 更费时、费力 | - | [ | |
| 扩增片段长度多态性结合一代测序技术 | 利用限制性内切酶水解基因组DNA,再使双链人工接头的酶切片段相连接,作为扩增反应的模板DNA,接头的互补链为引物进行预扩增,通过聚丙烯酰胺凝胶电泳分离检测 | 分辨率高、稳定性好、效率高 | 费用高,对DNA的纯度和内切酶的质量要求很高 | - | [ | |
| 临界温度退火PCR | PCR中临界退火温度抑制引物与模板的不匹配退火,抑制扩增子产生 | 简单、快速、经济,能够检测纯合突变 | 需要设计特异引物耗时费力 | 20%,1 bp | [ | |
| 特异性末端介导的聚合酶链式反应 | 设计产生带有特定末端部分的分子构建体,根据是否能够自折叠形成引物启动PCR扩增定性定量鉴定 | 能够定性定量检测单碱基突变,成本低、快速 | 引物设计复杂 | 30拷贝·反应-1 | [ | |
| 基于qPCR方法 | 实时荧光PCR和标记的实验 | 针对突变位点设计不同修饰类型探针,结合qPCR技术 | 简单、有效、灵敏 | 不能明确突变中核酸的变化 | <10%,1 bp | [ |
| 高分辨率熔解曲线分析技术 | 纯合DNA具有独特的退火温度,而突变的杂合DNA具有较低的熔化温度 | 快速有效检测SNP和indel类型的编辑 | 需要特殊的仪器,灵敏度受扩增产物长度影响 | 4.7%,2 bp | [ | |
| 基于聚合酶链式反应的高分辨片段分析方法 | 通过设计大小不同并用不同荧光团标记的PCR扩增子来对具有一个以上靶向基因的样品进行多重分析 | 灵敏度高,可检测到小至1 bp,能够区分多个突变的等位基因 | 大小相同的indel等位不能共同被洗脱和分辨 | 2%,1 bp | [ | |
| 简单等位基因辨别聚合酶链式反应 | 利用一种位点特异性反向引物,通用荧光探针和猝灭剂,以及热启动DNA聚合酶 | 快速、精准、低成本 | 需要提前优化PCR反应条件 | 0.4 ng·10 μL-1 | [ | |
| 基于数字PCR方法 | 数字PCR方法 | 将样品分割到许多单独的qPCR反应中,阴性结果用于生成样品中目标分子精确数量的绝对结果,无须参考标准品或内源性对照品 | 精准、低含量核酸加工食品、多倍体 | 成本高,需要特殊设备,不适用于大片段突变 | 1/100 000 | [ |
| 基于测序技术 | 全基因组测序 | 建库进行高通量测序 | 鉴定突变打靶、打靶类型和脱靶 | 成本高、耗时,可能存在内生菌或病原物序列信息的污染等 | - | [ |
| Sanger测序技术 | 第一代测序技术 | 准确鉴定突变位点 | 成本高、耗时、费力 | - | [ | |
| 基于其他技术 | CRISPR/Cas系统 | 利用CRISPR/Cas12特异性识别编辑位点和非特异性切割的功能 | 低成本、条件温和、操作简单、灵敏度高、能实现快速检测 | 可能存在脱靶效应导致假阳性或假阴性,需要PAM/PFS识别序列,难以标准化,需要扩增过程 | 12 拷贝·μL-1,1 bp | [ |
| 1 | SPRINK T, WILHELM R, HARTUNG F. Genome editing around the globe: an update on policies and perceptions[J]. Plant Physiol., 2022, 190(3): 1579-1587. |
| 2 | NAGAMINE A, EZURA H. Genome editing for improving crop nutrition[J/OL]. Front. Genome Ed., 2022, 4: 850104[2024-07-25]. . |
| 3 | KANG Y, DENG H, PRAY C, et al.. Managers' attitudes toward gene-editing technology and companies' R&D investment in gene-editing: the case of Chinese seed companies[J]. GM Crops Food, 2022, 13(1): 309-326. |
| 4 | 李欣,刘旭霞,张文斐.全球农业基因编辑技术监管动态及发展趋势[J].生命科学,2023,35(2):114-122. |
| LI X, LIU X X, ZHANG W F. Supervision dynamic and development tendency of global agricultural gene editing technology[J]. Chin. Bull. Life Sci., 2023, 35(2): 114-122. | |
| 5 | MANGHWAR H, LI B, DING X, et al.. CRISPR/Cas systems in genome editing: methodologies and tools for sgRNA design, off-target evaluation, and strategies to mitigate off-target effects[J/OL]. Adv. Sci. (Weinh), 2020, 7(6): 1902312[2024-07-25]. . |
| 6 | DHUGGA K S. Gene editing to accelerate crop breeding[J/OL]. Front. Plant Sci., 2022, 13: 889995[2024-07-25]. . |
| 7 | TYUMENTSEVA M, TYUMENTSEV A, AKIMKIN V. CRISPR/Cas9 landscape: current state and future perspectives[J/OL]. Int. J. Mol. Sci., 2023, 24(22): 16077[2024-07-25]. . |
| 8 | SHAMSHIRGARAN Y, LIU J, SUMER H, et al.. Tools for efficient genome editing: ZFN, TALEN, and CRISPR[J]. Meth. Mol. Biol. Clifton N.J., 2022, 2495: 29-46. |
| 9 | KIM Y G, CHA J, CHANDRASEGARAN S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain[J]. Proc. Natl. Acad. Sci. USA, 1996, 93(3): 1156-1160. |
| 10 | MILLER J C, TAN S, QIAO G, et al.. A TALE nuclease architecture for efficient genome editing[J]. Nat. Biotechnol., 2011, 29(2): 143-148. |
| 11 | SAKUMA T, YAMAMOTO T. Updated overview of TALEN construction systems[J]. Meth. Mol. Biol., 2023, 2637: 27-39. |
| 12 | LI B, SUN C, LI J, et al.. Targeted genome-modification tools and their advanced applications in crop breeding[J]. Nat. Rev. Genet., 2024, 25: 603-622. |
| 13 | XUE C, GREENE E C. DNA repair pathway choices in CRISPR-Cas9-mediated genome editing[J]. Trends Genet., 2021, 37(7): 639-656. |
| 14 | LANDER E S. The heroes of CRISPR[J]. Cell, 2016, 164(1-2): 18-28. |
| 15 | CONG L, RAN F A, COX D, et al.. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121): 819-823. |
| 16 | SON S, PARK S R. Challenges facing CRISPR/Cas9-based genome editing in plants[J/OL]. Front. Plant Sci., 2022, 13: 902413[2024-07-25]. . |
| 17 | PARK J, CHOE S. DNA-free genome editing with preassembled CRISPR/Cas9 ribonucleoproteins in plants[J]. Transgenic Res., 2019, 28(): 61-64. |
| 18 | ZHANG Y, MA X, XIE X, et al.. CRISPR/Cas9-based genome editing in plants[J]. Prog. Mol. Biol. Transl. Sci., 2017, 149: 133-150. |
| 19 | PANDEY P K, QUILICHINI T D, VAID N, et al.. Versatile and multifaceted CRISPR/Cas gene editing tool for plant research[J]. Semin. Cell Dev. Biol., 2019, 96: 107-114. |
| 20 | LIU Z, SCHIEL J A, MAKSIMOVA E, et al.. ErCas12a CRISPR-MAD7 for model generation in human cells, mice, and rats[J]. CRISPR J., 2020, 3(2): 97-108. |
| 21 | LIU Z X, ZHANG S, ZHU H Z, et al.. Hydrolytic endonucleolytic ribozyme (HYER) is programmable for sequence-specific DNA cleavage[J/OL]. Science, 2024, 383(6682): eadh4859[2024-07-25]. . |
| 22 | ALTAE-TRAN H, KANNAN S, DEMIRCIOGLU F E, et al.. The widespread IS200/IS605 transposon family encodes diverse programmable RNA-guided endonucleases[J]. Science, 2021, 374(6563): 57-65. |
| 23 | KARVELIS T, DRUTEIKA G, BIGELYTE G, et al.. Transposon-associated TnpB is a programmable RNA-guided DNA endonuclease[J]. Nature, 2021, 599(7886): 692-696. |
| 24 | HU J, SUN Y, LI B, et al.. Strand-preferred base editing of organellar and nuclear genomes using CyDENT[J]. Nat. Biotechnol., 2024, 42(6): 936-945. |
| 25 | DUAN Z, LIANG Y, SUN J, et al.. An engineered Cas12i nuclease that is an efficient genome editing tool in animals and plants[J/OL]. Innovation (Camb), 2024, 5(2): 100564[2024-07-25]. . |
| 26 | XIN C, QIAO D, WANG J, et al.. Enhanced editing efficiency in Arabidopsis with a LbCas12a variant harboring D156R and E795L mutations[J]. aBiotech, 2024, 5(2): 117-126. |
| 27 | LI W, LI X, WANG C, et al.. Expanding the targeting scope of CRISPR/Cas9-mediated genome editing by Cas9 variants in Brassica [J]. aBiotech, 2024, 5(2): 202-208. |
| 28 | GAO C. Genome engineering for crop improvement and future agriculture[J]. Cell, 2021, 184(6): 1621-1635. |
| 29 | KAZAMA T, OKUNO M, WATARI Y, et al.. Curing cytoplasmic male sterility via TALEN-mediated mitochondrial genome editing[J]. Nat. Plants, 2019, 5(7): 722-730. |
| 30 | MOK B Y, DE MORAES M H, ZENG J, et al.. A bacterial cytidine deaminase toxin enables CRISPR-free mitochondrial base editing[J]. Nature, 2020, 583(7817): 631-637. |
| 31 | YI Z, ZHANG X, TANG W, et al.. Strand-selective base editing of human mitochondrial DNA using mitoBEs[J]. Nat. Biotechnol., 2024, 42(3): 498-509. |
| 32 | WANG M, WANG H, LI K, et al.. Review of CRISPR/cas systems on detection of nucleotide sequences[J/OL]. Foods, 2023, 12(3): 477[2024-07-25]. . |
| 33 | 潘志文,张旭冬,高洁儿,等.基因组编辑植物的监管与检测技术[J].科技导报,2021,39(9):87-92. |
| PAN Z W, ZHANG X D, GAO J E, et al.. Supervision and detection technology of genome-edited plants[J]. Sci. Technol. Rev., 2021, 39(9): 87-92. | |
| 34 | KOMOR A C, KIM Y B, PACKER M S, et al.. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage[J]. Nature, 2016, 533(7603): 420-424. |
| 35 | NISHIDA K, ARAZOE T, YACHIE N, et al.. Targeted nucleotide editing using hybrid prokaryotic and vertebrate adaptive immune systems[J/OL]. Science, 2016, 353(6305): aaf8729[2024-07-25]. . |
| 36 | TONG H, WANG X, LIU Y, et al.. Programmable A-to-Y base editing by fusing an adenine base editor with an N-methylpurine DNA glycosylase[J]. Nat. Biotechnol., 2023, 41(8): 1080-1084. |
| 37 | CHEN L, HONG M, LUAN C, et al.. Adenine transversion editors enable precise, efficient A·T-to-C·G base editing in mammalian cells and embryos[J]. Nat. Biotechnol., 2024, 42(4): 638-650. |
| 38 | WU X, REN B, LIU L, et al.. Adenine base editor incorporating the N-methylpurine DNA glycosylase MPGv3 enables efficient A-to-K base editing in rice[J/OL]. Plant Commun., 2023, 4(6): 100668[2024-07-25]. . |
| 39 | LI Y, LI S, LI C, et al.. Fusion of a rice endogenous N-methylpurine DNA glycosylase to a plant adenine base transition editor ABE8e enables A-to-K base editing in rice plants[J]. aBIOTECH, 2024, 5(2): 127-139. |
| 40 | TONG H, LIU N, WEI Y, et al.. Programmable deaminase-free base editors for G-to-Y conversion by engineered glycosylase[J/OL]. Natl. Sci. Rev., 2023, 10(8): nwad143[2024-07-25]. . |
| 41 | HE Y, ZHOU X, CHANG C, et al.. Protein language models-assisted optimization of a uracil-N-glycosylase variant enables programmable T-to-G and T-to-C base editing[J]. Mol. Cell, 2024, 84(7): 1257-1270.e6. |
| 42 | 李欢欢, 宋馨, 夏永军, 等. 新型基因组编辑技术碱基编辑器研究进展[J/OL]. 食品与发酵工业, 2023, 038859[2024-07-25]. . |
| LI H H, SONG X, XIA Y J, et al.. Research progress of novel genome editing technology base editor [J]. Food Ferment Ind., 2023[2024-07-25]. . | |
| 43 | YI Z, QU L, TANG H, et al.. Engineered circular ADAR-recruiting RNAs increase the efficiency and fidelity of RNA editing in vitro and in vivo [J]. Nat. Biotechnol., 2022, 40(6): 946-955. |
| 44 | ANZALONE A V, RANDOLPH P B, DAVIS J R, et al.. Search-and-replace genome editing without double-strand breaks or donor DNA[J]. Nature, 2019, 576(7785): 149-157. |
| 45 | WANG S, ZONG Y, LIN Q, et al.. Precise, predictable multi-nucleotide deletions in rice and wheat using APOBEC-Cas9 [J]. Nat. Biotechnol., 2020, 38(12): 1460-1465. |
| 46 | SUN C, LEI Y, LI B, et al.. Precise integration of large DNA sequences in plant genomes using PrimeRoot editors[J]. Nat. Biotechnol., 2024, 42(2): 316-327. |
| 47 | WANG H, ZHANG D, CHEN M, et al.. Genome editing of 3' UTR-embedded inhibitory region enables generation of gene knock-up alleles in plants[J/OL]. Plant Commun., 2024, 5(3): 100745[2024-07-25]. . |
| 48 | LAKSHMI JAYARAJ K, THULASIDHARAN N, ANTONY A, et al.. Targeted editing of tomato carotenoid isomerase reveals the role of 5' UTR region in gene expression regulation[J]. Plant Cell Rep., 2021, 40(4): 621-635. |
| 49 | LIU M, ZHANG X, XU W, et al.. Efficient and precise genomic deletion in rice using enhanced prime editing[J]. aBiotech, 2024, 5(2): 214-218. |
| 50 | EFSA G P, NAEGELI H, BRESSON J L, et al.. Applicability of the EFSA Opinion on site-directed nucleases type 3 for the safety assessment of plants developed using site-directed nucleases type 1 and 2 and oligonucleotide-directed mutagenesis[J/OL]. EFSA J. Eur. Food Saf. Auth., 2020, 18(11): e06299[2024-07-25]. . |
| 51 | YUE J J, HONG C-Y, WEI P, et al.. How to start your monocot CRISPR/Cas project: plasmid design, efficiency detection, and offspring analysis[J/OL]. Rice, 2020, 13(1): 9[2024-07-25]. . |
| 52 | SHAN Q, WANG Y, LI J, et al.. Genome editing in rice and wheat using the CRISPR/Cas system[J]. Nat. Protoc., 2014, 9(10): 2395-2410. |
| 53 | HODGENS C, NIMCHUK Z L, KIEBER J J. IndCAPS: a tool for designing screening primers for CRISPR/Cas9 mutagenesis events[J/OL]. PLoS One, 2017, 12(11): e0188406[2024-07-25]. . |
| 54 | VOUILLOT L, THÉLIE A, POLLET N. Comparison of T7E1 and surveyor mismatch cleavage assays to detect mutations triggered by engineered nucleases[J]. G3 (Bethesda), 2015, 5(3): 407-415. |
| 55 | KC R, SRIVASTAVA A, WILKOWSKI J M, et al.. Detection of nucleotide-specific CRISPR/Cas9 modified alleles using multiplex ligation detection[J/OL]. Sci. Rep., 2016, 6: 32048[2024-07-25]. . |
| 56 | FOSTER S D, GLOVER S R, TURNER A N, et al.. A mixing heteroduplex mobility assay (mHMA) to genotype homozygous mutants with small indels generated by CRISPR-Cas9 nucleases[J]. MethodsX, 2019, 6: 1-5. |
| 57 | ZHENG X, YANG S, ZHANG D, et al.. Effective screen of CRISPR/Cas9-induced mutants in rice by single-strand conformation polymorphism[J]. Plant Cell Rep., 2016, 35(7): 1545-1554. |
| 58 | ZHU X, XU Y, YU S, et al.. An efficient genotyping method for genome-modified animals and human cells generated with CRISPR/Cas9 system[J/OL]. Sci. Rep., 2014, 4: 6420[2024-07-25]. . |
| 59 | LU Y, ZHU J K. Precise editing of a target base in the rice genome using a modified CRISPR/Cas9 system[J]. Mol. Plant, 2017, 10(3): 523-525. |
| 60 | DENG L, WANG H, SUN C, et al.. Efficient generation of pink-fruited tomatoes using CRISPR/Cas9 system[J]. J. Genet. Genomics, 2018, 45(1): 51-54. |
| 61 | THOMAS H R, PERCIVAL S M, YODER B K, et al.. High-throughput genome editing and phenotyping facilitated by high resolution melting curve analysis[J/OL]. PLoS One, 2014, 9(12): e114632[2024-07-25]. . |
| 62 | XU G, YANG H, QIU J, et al.. Sequence terminus dependent PCR for site-specific mutation and modification detection[J/OL]. Nat. Commun., 2023, 14(1): 1169[2024-07-25]. . |
| 63 | CHHALLIYIL P, ILVES H, KAZAKOV S A, et al.. A real-time quantitative PCR method specific for detection and quantification of the first commercialized genome-edited plant[J/OL]. Foods, 2020, 9(9): 1245[2024-07-25]. . |
| 64 | PENG C, WANG H, XU X, et al.. High-throughput detection and screening of plants modified by gene editing using quantitative real-time polymerase chain reaction[J]. Plant J., 2018, 95(3): 557-567. |
| 65 | M-AFRAITURE, D'AES J, GUIDERDONI E, et al.. Targeted high-throughput sequencing enables the detection of single nucleotide variations in CRISPR/Cas9 gene-edited organisms[J/OL]. Foods, 2023, 12(3): 455[2024-07-25]. . |
| 66 | ANDERSSON M, TURESSON H, NICOLIA A, et al.. Efficient targeted multiallelic mutagenesis in tetraploid potato (Solanum tuberosum) by transient CRISPR-Cas9 expression in protoplasts[J]. Plant Cell Rep., 2017, 36(1): 117-128. |
| 67 | BUI M, LIU Z. Simple allele-discriminating PCR for cost-effective and rapid genotyping and mapping[J/OL]. Plant Methods, 2009, 5: 1[2024-07-25]. . |
| 68 | TANG X, LIU G, ZHOU J, et al.. A large-scale whole-genome sequencing analysis reveals highly specific genome editing by both Cas9 and Cpf1 (Cas12a) nucleases in rice[J/OL]. Genome Biol., 2018, 19(1): 84[2024-07-25]. . |
| 69 | WANG M, LIU X, YANG J, et al.. CRISPR/Cas12a-based biosensing platform for the on-site detection of single-base mutants in gene-edited rice[J/OL]. Front. Plant Sci., 2022, 13: 944295[2024-07-25]. . |
| 70 | HUA Y, WANG C, HUANG J, et al.. A simple and efficient method for CRISPR/Cas9-induced mutant screening[J]. Yi Chuan Xue Bao, 2017, 44(4): 207-213. |
| 71 | PENG C, ZHENG M, DING L, et al.. Accurate detection and evaluation of the gene-editing frequency in plants using droplet digital PCR[J/OL]. Front. Plant Sci., 2020, 11: 610790[2024-07-25]. . |
| 72 | GAO R, FEYISSA B A, CROFT M, et al.. Gene editing by CRISPR/Cas9 in the obligatory outcrossing Medicago sativa . |
| 73 | BISWAS S, LI R, YUAN Z, et al.. Development of methods for effective identification of CRISPR/Cas9-induced indels in rice[J]. Plant Cell Rep., 2019, 38(4): 503-510. |
| 74 | MA X, CHEN L, ZHU Q, et al.. Rapid decoding of sequence-specific nuclease-induced heterozygous and biallelic mutations by direct sequencing of PCR products[J]. Mol. Plant, 2015, 8(8): 1285-1287. |
| 75 | PENG C, ZHENG M, DING L, et al.. Accurate detection and evaluation of the gene-editing frequency in plants using droplet digital PCR[J/OL]. Front. Plant Sci., 2020, 11: 610790[2024-07-25]. . |
| 76 | LIU Q, WANG C, JIAO X, et al.. Hi-TOM: a platform for high-throughput tracking of mutations induced by CRISPR/Cas systems[J]. Sci. China Life Sci., 2019, 62(1): 1-7. |
| 77 | BHUYAN S J, KUMAR M, RAMRAO DEVDE P, et al.. Progress in gene editing tools, implications and success in plants: a review[J/OL]. Front. Genome Ed., 2023, 5: 1272678[2024-07-25]. . |
| 78 | 丁霖,王颢潜,张敏,等.基因编辑外源基因Cas9定性定量检测方法的研究[J].农业生物技术学报,2022,30(10):1855-1868. |
| DING L, WANG H Q, ZHANG M, et al.. Study of qualitative and quantitative detection methods for exogenous gene Cas9 in gene editing[J]. J. Agric. Biotechnol., 2022, 30(10): 1855-1868. | |
| 79 | SPRINK T, ERIKSSON D, SCHIEMANN J, et al.. Regulatory hurdles for genome editing: process-vs. product-based approaches in different regulatory contexts[J]. Plant Cell Rep., 2016, 35(7): 1493-1506. |
| 80 | AHMAD A, MUNAWAR N, KHAN Z, et al.. An outlook on global regulatory landscape for genome-edited crops[J/OL]. Int. J. Mol. Sci., 2021, 22(21): 11753[2024-07-25]. . |
| 81 | GUPTA S, KUMAR A, PATEL R, et al.. Genetically modified crop regulations: scope and opportunity using the CRISPR-Cas9 genome editing approach[J]. Mol. Biol. Rep., 2021, 48(5): 4851-4863. |
| 82 | 张丽雯,刘加兰,王洪,等.基因编辑技术监管现状研究[J].生命科学,2022,34(10):1317-1326. |
| ZHANG L W, LIU J L, WANG H, et al.. Current supervision status of gene editing technology[J]. Chin. Bull. Life Sci., 2022, 34(10): 1317-1326. | |
| 83 | PUCHTA H. Regulation of gene-edited plants in Europe: from the valley of tears into the shining Sun?[J]. aBiotech, 2023, 5(2): 231-238. |
| [1] | 贾名扬, 王磊, 陈俊峰, 张家庆, 闫祥洲, 邢宝松, 王璟. CRISPR/Cas9基因编辑技术在畜禽育种中的研究进展[J]. 生物技术进展, 2024, 14(4): 529-536. |
| [2] | 张笑天, 王智, 朱鹏宇, 魏霜, 付伟, 黄春蒙, 李志红, 王慧煜, 焦悦. 一种基于定量PCR的CRISPR/Cas9基因编辑作物快速检测方法的研究[J]. 生物技术进展, 2023, 13(6): 907-912. |
| [3] | 曹克浩, 朱军利, 何华山, 徐慰倬. 专利法第四次修改对生物技术专利申请和产业发展的影响[J]. 生物技术进展, 2023, 13(5): 663-670. |
| [4] | 王阿利, 刘江东. CRISPR/Cas系统在斑马鱼中的研究进展[J]. 生物技术进展, 2023, 13(4): 485-491. |
| [5] | 盖思宇, 陈子奇, 夏涵超, 赵仁贵, 刘相国. 基因编辑技术在植物启动子编辑中的研究进展[J]. 生物技术进展, 2023, 13(3): 321-328. |
| [6] | 孙卉, 张春义, 姜凌. 药用植物分子农场研究进展[J]. 生物技术进展, 2023, 13(1): 65-71. |
| [7] | 杨洋, 王凤林, 刘德, 罗园园, 朱建华. CRISPR⁃Cas9技术在植物次生代谢物生产中的研究进展[J]. 生物技术进展, 2022, 12(6): 806-816. |
| [8] | 于鲲, 薛佳琪, 王进宽, 余永涛. CRISPR/Cas9基因编辑技术在丝状真菌中的应用[J]. 生物技术进展, 2022, 12(5): 696-704. |
| [9] | 刘婷婷, 仝涛, 黄昆仑. 转基因玉米的研究进展和食用安全性评价[J]. 生物技术进展, 2022, 12(4): 523-531. |
| [10] | 高维崧, 窦金萍, 韦双, 刘兴健, 张志芳, 李轶女. CRISPR/Cas系统的分类及研究现状[J]. 生物技术进展, 2022, 12(4): 532-538. |
| [11] | 党星, 郅斌伟, 曹克浩, 刘婷婷, 陈飚, 丁远杰. 转基因玉米生物育种技术的专利分析及产业发展建议[J]. 生物技术进展, 2022, 12(4): 614-622. |
| [12] | 费云燕, 杨军, 景德道, 林添资, 李闯, 钱华飞, 曾生元, 韩华新, 龚红兵. CRISPR/Cas技术在抗除草剂作物育种中的研究与应用进展[J]. 生物技术进展, 2022, 12(2): 189-197. |
| [13] | 黄耀辉, 王艺洁, 杨立桃, 焦悦, 付仲文. 生物育种新技术作物的安全管理[J]. 生物技术进展, 2022, 12(2): 198-204. |
| [14] | 曹巧, 史占良, 张国丛, 班进福, 郑树松, 傅晓艺, 张士昌, 何明琦, 韩然, 高振贤. CRISPR/Cas9技术在小麦育种中的应用进展[J]. 生物技术进展, 2021, 11(6): 661-667. |
| [15] | 林敏. 农业生物育种技术的发展历程及产业化对策[J]. 生物技术进展, 2021, 11(4): 405-417. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||
版权所有 © 2021《生物技术进展》编辑部