


生物技术进展 ›› 2024, Vol. 14 ›› Issue (3): 433-441.DOI: 10.19586/j.2095-2341.2023.0162
• 研究论文 • 上一篇
收稿日期:2023-12-14
接受日期:2024-01-24
出版日期:2024-05-25
发布日期:2024-06-18
通讯作者:
李凤梅,崔健
作者简介:张慧 E-mail: 824572941@qq.com;
基金资助:
Hui ZHANG1( ), Bobo LIU1, Fengmei LI1(
), Bobo LIU1, Fengmei LI1( ), Jian CUI2(
), Jian CUI2( )
)
Received:2023-12-14
Accepted:2024-01-24
Online:2024-05-25
Published:2024-06-18
Contact:
Fengmei LI,Jian CUI
摘要:
生长素(auxin, IAA)信号途径在植物生长、生物胁迫和非生物胁迫响应中发挥着重要的作用。白粉病是南瓜普遍发生且较为严重的一种病害,为探究IAA信号途径响应白粉病胁迫的分子机制,对白粉菌处理的南瓜叶片进行了转录组测序和全基因组DNA甲基化测序分析。结果发现,在IAA信号途径中,有25个差异表达基因,53个基因发生了差异甲基化,其中16个生长素上调小RNA (SAUR)基因均发生了不同程度的甲基化,说明这些基因可能参与了白粉病胁迫响应。SAUR50(CmoCh19G007170)基因的甲基化水平降低,且甲基化区域位于基因的启动子区。在白粉病胁迫下SAUR50的表达水平显著上调,在IAA诱导下显著下调,因此该基因可能通过DNA甲基化调节其表达水平,并且通过IAA信号途径参与南瓜白粉病胁迫的调控。研究结果为IAA信号途径响应白粉病胁迫途径与抗白粉病南瓜分子育种提供了理论依据。
中图分类号:
张慧, 刘愽愽, 李凤梅, 崔健. 生长素信号途径参与南瓜白粉菌胁迫的响应研究[J]. 生物技术进展, 2024, 14(3): 433-441.
Hui ZHANG, Bobo LIU, Fengmei LI, Jian CUI. Study on the Involvement of Auxin Signaling Pathway in Response to Powdery Mildew Stress in Pumpkin[J]. Current Biotechnology, 2024, 14(3): 433-441.
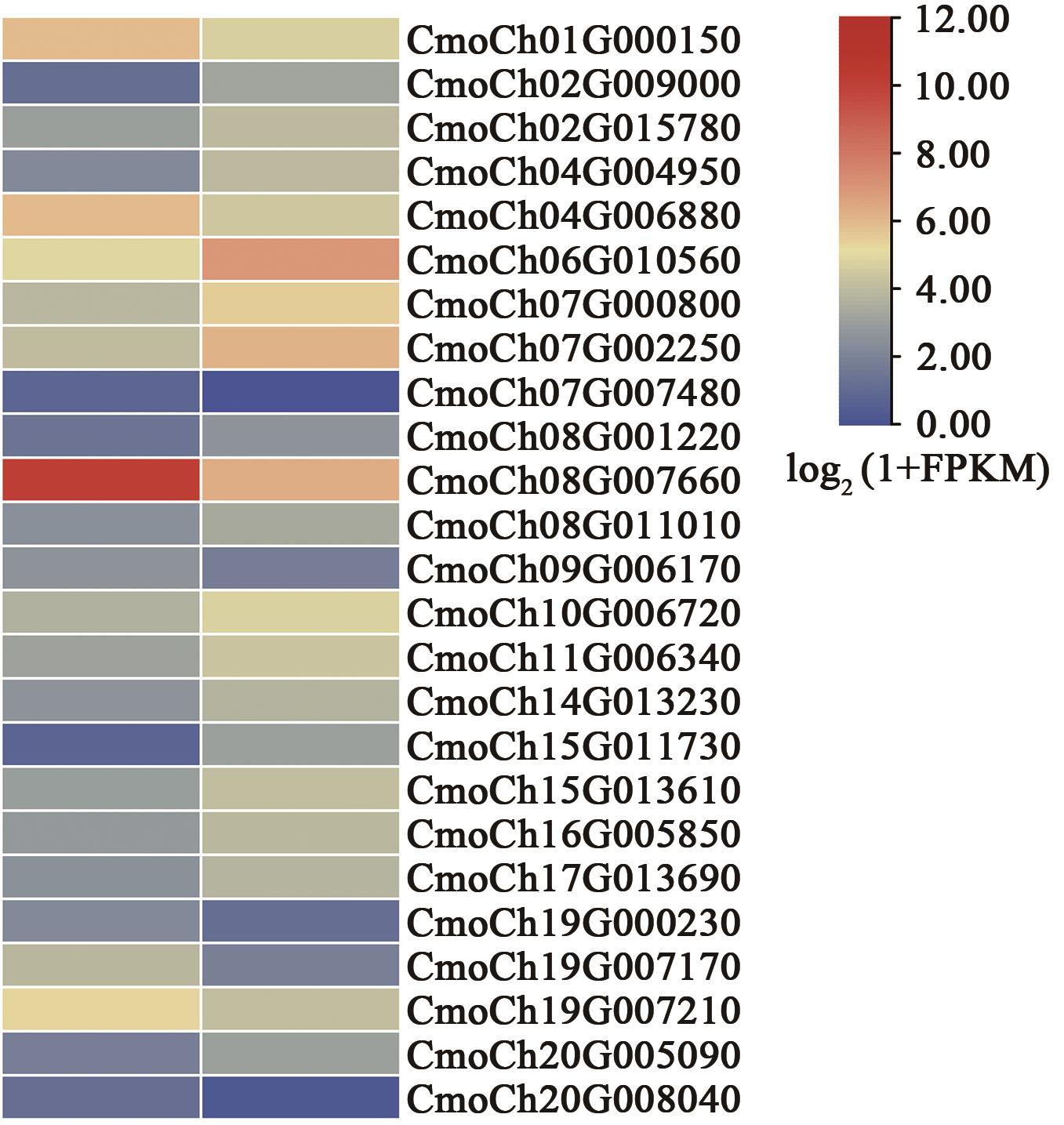
图1 白粉病处理后IAA信号途径相关基因表达热图注:每列代表感染白粉病和未感染白粉病南瓜叶片中3个生物学重复的log2(FKPM+1)平均值。
Fig. 1 Heat map of gene expression related to auxin signaling pathway after treatment with powdery mildew
| 基因 ID | log2|FoldChange| | 基因起始位点 | 基因终止位点 | 注释 |
|---|---|---|---|---|
| CmoCh01G000150 | 1.192 986 569 | 51 481 | 52 456 | 生长素相关蛋白 |
| CmoCh02G009000 | -2.729 158 581 | 5 550 636 | 5 554 600 | 生长素应答因子22 |
| CmoCh02G015780 | -1.085 102 287 | 9 109 056 | 9 112 669 | 生长素渠化 |
| CmoCh04G004950 | -2.026 235 227 | 2 470 212 | 2 472 488 | 生长素响应GH3样蛋白1 |
| CmoCh04G006880 | 1.535 353 461 | 3 408 834 | 3 412 302 | 生长素诱导蛋白 |
| CmoCh06G010560 | -2.044 779 387 | 8 210 781 | 8 214 108 | 生长素响应GH3样蛋白5 |
| CmoCh07G000800 | -1.672 989 912 | 500 794 | 505 922 | 生长素应答因子 |
| CmoCh07G002250 | -2.181 053 727 | 1 136 983 | 1 140 904 | 生长素抑制蛋白 |
| CmoCh07G007480 | 3.270 328 289 | 3 388 548 | 3 390 222 | 生长素应答因子36 |
| CmoCh08G001220 | -1.678 882 862 | 629 552 | 633 268 | 生长素反应蛋白IAA26 |
| CmoCh08G007660 | 3.895 043 450 | 4 840 255 | 4 840 875 | 生长素结合蛋白 |
| CmoCh08G011010 | -1.027 143 495 | 7 056 498 | 7 063 138 | 生长素渠化 |
| CmoCh09G006170 | 1.054 262 364 | 3 084 770 | 3 091 533 | 生长素渠化 |
| CmoCh10G006720 | -1.210 110 125 | 3 087 573 | 3 089 725 | 生长素反应蛋白IAA27 |
| CmoCh11G006340 | -1.374 547 937 | 3 047 847 | 3 051 459 | 生长素反应蛋白IAA27 |
| CmoCh14G013230 | -1.248 495 558 | 11 047 643 | 11 049 773 | 生长素反应蛋白IAA26 |
| CmoCh15G011730 | -3.433 307 646 | 8 165 466 | 8 169 541 | 生长素渠化 |
| CmoCh15G013610 | -1.282 714 907 | 9 303 666 | 9 310 054 | 生长素应答因子9 |
| CmoCh16G005850 | -1.222 754 998 | 2 822 610 | 2 826 352 | 生长素应答因子9 |
| CmoCh17G013690 | -1.441 604 240 | 10 631 575 | 10 632 190 | 生长素反应蛋白 |
| CmoCh19G000230 | 1.584 356 252 | 139 153 | 141 383 | 生长素渠化 |
| CmoCh19G007170 | 2.359 833 611 | 7 501 456 | 7 501 755 | 生长素反应蛋白SAUR50 |
| CmoCh19G007210 | 1.222 892 554 | 7 514 737 | 7 515 027 | 生长素诱导蛋白 |
| CmoCh20G005090 | -1.540 231 132 | 2 433 984 | 2 440 115 | 生长素应答因子4 |
| CmoCh20G008040 | 2.795 275 878 | 3 960 038 | 3 962 188 | 生长素反应蛋白SAUR24 |
表1 IAA信号途径相关基因差异表达水平及注释信息
Table 1 Differential expression levels and annotation information of genes related to the IAA signaling pathway
| 基因 ID | log2|FoldChange| | 基因起始位点 | 基因终止位点 | 注释 |
|---|---|---|---|---|
| CmoCh01G000150 | 1.192 986 569 | 51 481 | 52 456 | 生长素相关蛋白 |
| CmoCh02G009000 | -2.729 158 581 | 5 550 636 | 5 554 600 | 生长素应答因子22 |
| CmoCh02G015780 | -1.085 102 287 | 9 109 056 | 9 112 669 | 生长素渠化 |
| CmoCh04G004950 | -2.026 235 227 | 2 470 212 | 2 472 488 | 生长素响应GH3样蛋白1 |
| CmoCh04G006880 | 1.535 353 461 | 3 408 834 | 3 412 302 | 生长素诱导蛋白 |
| CmoCh06G010560 | -2.044 779 387 | 8 210 781 | 8 214 108 | 生长素响应GH3样蛋白5 |
| CmoCh07G000800 | -1.672 989 912 | 500 794 | 505 922 | 生长素应答因子 |
| CmoCh07G002250 | -2.181 053 727 | 1 136 983 | 1 140 904 | 生长素抑制蛋白 |
| CmoCh07G007480 | 3.270 328 289 | 3 388 548 | 3 390 222 | 生长素应答因子36 |
| CmoCh08G001220 | -1.678 882 862 | 629 552 | 633 268 | 生长素反应蛋白IAA26 |
| CmoCh08G007660 | 3.895 043 450 | 4 840 255 | 4 840 875 | 生长素结合蛋白 |
| CmoCh08G011010 | -1.027 143 495 | 7 056 498 | 7 063 138 | 生长素渠化 |
| CmoCh09G006170 | 1.054 262 364 | 3 084 770 | 3 091 533 | 生长素渠化 |
| CmoCh10G006720 | -1.210 110 125 | 3 087 573 | 3 089 725 | 生长素反应蛋白IAA27 |
| CmoCh11G006340 | -1.374 547 937 | 3 047 847 | 3 051 459 | 生长素反应蛋白IAA27 |
| CmoCh14G013230 | -1.248 495 558 | 11 047 643 | 11 049 773 | 生长素反应蛋白IAA26 |
| CmoCh15G011730 | -3.433 307 646 | 8 165 466 | 8 169 541 | 生长素渠化 |
| CmoCh15G013610 | -1.282 714 907 | 9 303 666 | 9 310 054 | 生长素应答因子9 |
| CmoCh16G005850 | -1.222 754 998 | 2 822 610 | 2 826 352 | 生长素应答因子9 |
| CmoCh17G013690 | -1.441 604 240 | 10 631 575 | 10 632 190 | 生长素反应蛋白 |
| CmoCh19G000230 | 1.584 356 252 | 139 153 | 141 383 | 生长素渠化 |
| CmoCh19G007170 | 2.359 833 611 | 7 501 456 | 7 501 755 | 生长素反应蛋白SAUR50 |
| CmoCh19G007210 | 1.222 892 554 | 7 514 737 | 7 515 027 | 生长素诱导蛋白 |
| CmoCh20G005090 | -1.540 231 132 | 2 433 984 | 2 440 115 | 生长素应答因子4 |
| CmoCh20G008040 | 2.795 275 878 | 3 960 038 | 3 962 188 | 生长素反应蛋白SAUR24 |

图2 白粉病处理后IAA信号途径相关基因的差异甲基化热图注:每列表示感染白粉病和未感染白粉病南瓜叶片中3个生物学重复的平均甲基化水平。
Fig. 2 Differential methylation heatmap of genes to related auxin signaling pathway after powdery mildew treatment
| 基因ID | 甲基化类型 | 甲基化区域 | 基因ID | 甲基化类型 | 甲基化区域 |
|---|---|---|---|---|---|
| CmoCh01G010370 | CHH | 内含子 | CmoCh13G008530 | CHH | 启动子 |
| CmoCh02G003140 | CHH | 内含子 | CmoCh14G006160 | CHH | 内含子 |
| CmoCh03G012750 | CHH | 启动子 | CmoCh14G006150 | CHH | 内含子 |
| CmoCh04G007210 | CHH | 启动子 | CmoCh14G013230 | CHH | 内含子 |
| CmoCh04G029470 | CHH | 启动子 | CmoCh14G015800 | CHH | 启动子 |
| CmoCh04G030360 | CHH | 启动子 | CmoCh14G018090 | CHH | 启动子 |
| CmoCh05G007670 | CHH, CG | 内含子, 外显子 | CmoCh15G001380 | CHH | 内含子 |
| CmoCh05G008000 | CHH | 启动子 | CmoCh15G009810 | CHH | 启动子 |
| CmoCh07G002150 | CHH | 启动子 | CmoCh17G001350 | CHH | 启动子 |
| CmoCh07G009450 | CHH | 启动子 | CmoCh17G011930 | CG, CHH | 启动子 |
| CmoCh07G010990 | CHH | 启动子 | CmoCh18G004680 | CHH | 内含子 |
| CmoCh09G001100 | CHH | 启动子 | CmoCh18G010050 | CHH | 内含子 |
| CmoCh09G004620 | CHH | 内含子 | CmoCh19G001290 | CHH | 启动子 |
| CmoCh10G005700 | CHH | 启动子 | CmoCh19G007160 | CHH | 启动子 |
| CmoCh11G003520 | CG | 外显子 | CmoCh19G007170 | CHH | 启动子 |
| CmoCh11G007400 | CG | 启动子 | CmoCh19G007180 | CHH | 启动子 |
| CmoCh11G015800 | CHH | 启动子 | CmoCh19G007290 | CG | 启动子 |
| CmoCh11G017120 | CHH | 启动子 | CmoCh19G007400 | CHH | 内含子, 启动子 |
| CmoCh11G017130 | CHH | 启动子 | CmoCh20G005420 | CHH | 启动子 |
| CmoCh11G017140 | CHH | 启动子 | CmoCh20G007770 | CHH | 启动子 |
| CmoCh11G017180 | CHH | 启动子 | CmoCh20G007790 | CHH | 启动子 |
| CmoCh11G017330 | CG, CHG | 内含子, 外显子 | CmoCh20G007780 | CHH | 内含子 |
| CmoCh11G017660 | CHH | 启动子 | CmoCh20G008020 | CG | 启动子 |
| CmoCh12G001120 | CHH | 内含子, 启动子 | CmoCh20G008010 | CG | 启动子 |
| CmoCh12G003660 | CHH | 启动子 | CmoCh20G008070 | CHH | 启动子 |
| CmoCh12G011430 | CHH | 启动子 | CmoCh20G008080 | CHH | 启动子 |
| CmoCh12G013660 | CHH | 启动子 |
表2 IAA信号途径相关基因甲基化类型及区域
Table 2 Methylation types and regions of genes to related IAA signaling pathway
| 基因ID | 甲基化类型 | 甲基化区域 | 基因ID | 甲基化类型 | 甲基化区域 |
|---|---|---|---|---|---|
| CmoCh01G010370 | CHH | 内含子 | CmoCh13G008530 | CHH | 启动子 |
| CmoCh02G003140 | CHH | 内含子 | CmoCh14G006160 | CHH | 内含子 |
| CmoCh03G012750 | CHH | 启动子 | CmoCh14G006150 | CHH | 内含子 |
| CmoCh04G007210 | CHH | 启动子 | CmoCh14G013230 | CHH | 内含子 |
| CmoCh04G029470 | CHH | 启动子 | CmoCh14G015800 | CHH | 启动子 |
| CmoCh04G030360 | CHH | 启动子 | CmoCh14G018090 | CHH | 启动子 |
| CmoCh05G007670 | CHH, CG | 内含子, 外显子 | CmoCh15G001380 | CHH | 内含子 |
| CmoCh05G008000 | CHH | 启动子 | CmoCh15G009810 | CHH | 启动子 |
| CmoCh07G002150 | CHH | 启动子 | CmoCh17G001350 | CHH | 启动子 |
| CmoCh07G009450 | CHH | 启动子 | CmoCh17G011930 | CG, CHH | 启动子 |
| CmoCh07G010990 | CHH | 启动子 | CmoCh18G004680 | CHH | 内含子 |
| CmoCh09G001100 | CHH | 启动子 | CmoCh18G010050 | CHH | 内含子 |
| CmoCh09G004620 | CHH | 内含子 | CmoCh19G001290 | CHH | 启动子 |
| CmoCh10G005700 | CHH | 启动子 | CmoCh19G007160 | CHH | 启动子 |
| CmoCh11G003520 | CG | 外显子 | CmoCh19G007170 | CHH | 启动子 |
| CmoCh11G007400 | CG | 启动子 | CmoCh19G007180 | CHH | 启动子 |
| CmoCh11G015800 | CHH | 启动子 | CmoCh19G007290 | CG | 启动子 |
| CmoCh11G017120 | CHH | 启动子 | CmoCh19G007400 | CHH | 内含子, 启动子 |
| CmoCh11G017130 | CHH | 启动子 | CmoCh20G005420 | CHH | 启动子 |
| CmoCh11G017140 | CHH | 启动子 | CmoCh20G007770 | CHH | 启动子 |
| CmoCh11G017180 | CHH | 启动子 | CmoCh20G007790 | CHH | 启动子 |
| CmoCh11G017330 | CG, CHG | 内含子, 外显子 | CmoCh20G007780 | CHH | 内含子 |
| CmoCh11G017660 | CHH | 启动子 | CmoCh20G008020 | CG | 启动子 |
| CmoCh12G001120 | CHH | 内含子, 启动子 | CmoCh20G008010 | CG | 启动子 |
| CmoCh12G003660 | CHH | 启动子 | CmoCh20G008070 | CHH | 启动子 |
| CmoCh12G011430 | CHH | 启动子 | CmoCh20G008080 | CHH | 启动子 |
| CmoCh12G013660 | CHH | 启动子 |

图4 CmoCh19G007170(SAUR50)在不同处理下的相对表达水平A:白粉菌处理;B:IAA激素处理。不同小写字母表示处理间具有统计学差异(P<0.05)。
Fig. 4 Relative expression levels of CmoCh19G007170 (SAUR50) under different treatment
| 1 | PÉREZ-GARCÍA A, ROMERO D, FERNÁNDEZ-ORTUÑO D, et al.. The powdery mildew fungus Podosphaera fusca (synonym Podosphaera xanthii), a constant threat to cucurbits[J]. Mol. Plant Pathol., 2009, 10(2): 153-160. |
| 2 | BABADOOST M, SULLEY S, XIANG Y. Sensitivities of cucurbit powdery mildew fungus ( Podosphaera xanthii ) to fungicides[J]. Plant Hlth. Prog., 2020, 21:272-277. |
| 3 | BADRI A M, BAE I, LEE S. Marker-assisted evaluation of two powdery mildew resistance candidate genes in korean cucumber inbred lines[J]. Agronomy, 2021, 11(11):2191.?补 |
| 4 | 孙海,王晓青,李云龙,等.寡雄腐霉施用时期对设施黄瓜霜霉病的防治试验[J].生物技术进展,2014,4(1):63-65. |
| SUN H, WANG X Q, LI Y L, et al.. Evaluation of application time for Pythium oligandrum against cucumber downy mildew in greenhouse[J]. Curr. Biotechnol., 2014, 4(1): 63-65. | |
| 5 | 吴星波,郝俊杰,张晓艳,等.普通菜豆抗白粉病基因SCAR标记鉴定[J].生物技术进展,2013,3(5):357-362. |
| WU X B, HAO J J, ZHANG X Y, et al.. Identification of powdery mildew resistant genes based on SCAR markers in common bean (Phaseolus vulgaris L.)[J]. Curr. Biotechnol., 2013, 3(5): 357-362. | |
| 6 | 冯寒骞,李超.生长素信号转导研究进展[J].生物技术通报,2018,34(7):24-30. |
| FENG H Q, LI C. Research advances of auxin signal transduction[J]. Biotechnol. Bull., 2018, 34(7): 24-30. | |
| 7 | DHARMASIRI N, DHARMASIRI S, ESTELLE M. The F-box protein TIR1 is an auxin receptor[J]. Nature, 2005, 435: 441-445. |
| 8 | KEPINSKI S, LEYSER O. The Arabidopsis F-box protein TIR1 is an auxin receptor[J]. Nature, 2005, 435: 446-451. |
| 9 | TAN X, CALDERON-VILLALOBOS L I A, SHARON M, et al.. Mechanism of auxin perception by the TIR1 ubiquitin ligase[J]. Nature, 2007, 446: 640-645. |
| 10 | SALEHIN M, BAGCHI R, ESTELLE M. SCFTIR1/AFB-based auxin perception: mechanism and role in plant growth and development[J]. Plant Cell, 2015, 27(1): 9-19. |
| 11 | GRAY W M, KEPINSKI S, ROUSE D, et al.. Auxin regulates SCFTIR1-dependent degradation of AUX/IAA proteins[J]. Nature, 2001, 414: 271-276. |
| 12 | CHEN M, NIE G, YANG L, et al.. Homeotic transformation from stamen to petal in Lilium is associated with MADS-box genes and hormone signal transduction[J]. Plant Growth Regul., 2021, 95(1): 49-64. |
| 13 | JIN L, QIN Q, WANG Y, et al.. Rice dwarf virus P2 protein hijacks auxin signaling by directly targeting the rice OsIAA10 protein, enhancing viral infection and disease development[J/OL]. PLoS Pathog., 2016, 12(9): e1005847[2024-03-15]. . |
| 14 | MEHMOOD A, HUSSAIN A, IRSHAD M, et al.. IAA and flavonoids modulates the association between maize roots and phytostimulant endophytic Aspergillus fumigatus greenish[J]. J. Plant Interact., 2018, 13(1):532-542. |
| 15 | HE Y, ZHANG T, SUN Y, et al.. Exogenous IAA alleviates arsenic toxicity to rice and reduces arsenic accumulation in rice grains[J]. J. Plant Growth. Regul., 2022, 41(2): 734-741. |
| 16 | WEN T, DONG L, WANG L, et al.. Changes in root architecture and endogenous hormone levels in two Malus rootstocks under alkali stress[J]. Sci. Hortic. Amsterdam., 2018, 235:198-204. |
| 17 | NAVARRO L, DUNOYER P, JAY F, et al.. A plant miRNA contributes to antibacterial resistance by repressing auxin signaling[J]. Science, 2006, 312(5772): 436-439. |
| 18 | ZHANG Z, LI Q, LI Z, et al.. Dual regulation role of GH3.5 in salicylic acid and auxin signaling during Arabidopsis-Pseudomonas syringae interaction[J]. Plant Physiol., 2007, 145(2): 450-464. |
| 19 | PADMANABHAN M S, SHIFERAW H, CULVER J N. The Tobacco mosaic virus replicase protein disrupts the localization and function of interacting Aux/IAA proteins[J]. Mol. Plant Microbe Interact., 2006, 19(8): 864-873. |
| 20 | SUN Y, FAN M, HE Y. DNA methylation analysis of the Citrullus lanatus response to Cucumber GreenMottle mosaic virus infection by whole-genome bisulfite sequencing[J/OL]. Genes Basel., 2019, 10(5): e344[2024-03-15]. . |
| 21 | RAMBANI A, RICE J H, LIU J, et al.. The methylome of soybean roots during the compatible interaction with the soybean cyst nematode[J]. Plant Physiol., 2015, 168(4): 1364-1377. |
| 22 | 徐妍.灰斑病菌胁迫对大豆生理生化和DNA甲基化的影响[D].哈尔滨:哈尔滨师范大学,2015. |
| 23 | CAO X, ZHAI X, ZHAO Z, et al.. Genome-wide DNA methylation analysis of paulownia with phytoplasma infection[J/OL]. Gene, 2020, 755: 144905[2024-03-15]. . |
| 24 | WANG S, YAN W, YANG X, et al.. Comparative methylome reveals regulatory roles of DNA methylation in melon resistance to Podosphaera xanthii [J/OL]. Plant Sci., 2021, 309: 110954[2024-03-15]. . |
| 25 | ZITTER T A. Compendium of Cucurbit Diseases[M]. US: Amer Phytopathological Society, 1996. |
| 26 | 方汉顺,谢永盾,曾伟伟,等.小麦矮秆突变体jm22d响应赤霉素处理的转录组学分析[J].生物技术进展,2020,10(5):503-516. |
| FANG H S, XIE Y D, ZENG W W, et al.. The transcriptome analysis of wheat dwarf mutant jm22d responding to GA treatment[J]. Curr. Biotechnol., 2020, 10(5): 503-516. | |
| 27 | MO H J, SUN Y X, ZHU X L, et al.. Cotton S-adenosylmethionine decarboxylase-mediated spermine biosynthesis is required for salicylic acid- and leucine-correlated signaling in the defense response to Verticillium dahliae [J]. Planta, 2016, 243(4): 1023-1039. |
| 28 | PARK S Y, FUNG P, NISHIMURA N, et al.. Abscisic acid inhibits type 2C protein phosphatases via the PYR/PYL family of START proteins[J]. Science, 2009, 324(5930): 1068-1071. |
| 29 | WOODWARD A W, BARTEL B. Auxin: regulation, action, and interaction[J]. Ann. Bot., 2005, 95(5): 707-735. |
| 30 | 陈中惠,李景蕻.外源植物激素对高温胁迫下水培绿萝耐热性的影响[J].生物技术进展,2023,13(5):742-747. |
| CHEN Z H, LI J H. Effects of exogenous phytohormones on heat tolerance of Epipremnum aureum under hydroponic culture[J]. Curr. Biotechnol., 2023, 13(5): 742-747. | |
| 31 | LISCUM E, REED J W. Genetics of Aux/IAA and ARF action in plant growth and development[J]. Plant Mol. Biol., 2002, 49(3): 387-400. |
| 32 | HAGEN G, GUILFOYLE T. Auxin-responsive gene expression: genes, promoters and regulatory factors[J]. Plant Mol. Biol., 2002, 49(3-4): 373-385. |
| 33 | STASWICK P E, SERBAN B, ROWE M, et al.. Characterization of an Arabidopsis enzyme family that conjugates amino acids to indole-3-acetic acid [J]. Plant Cell, 2005, 17(2): 616-627. |
| 34 | NEWMAN T C, OHME-TAKAGI M, TAYLOR C B, et al.. DST sequences, highly conserved among plant SAUR genes, target reporter transcripts for rapid decay in tobacco[J]. Plant Cell, 1993, 5(6): 701-714. |
| 35 | LI Z G, CHEN H W, LI Q T, et al.. Three SAUR proteins SAUR 76, SAUR77 and SAUR78 promote plant growth in Arabidopsis[J/OL]. Sci. Rep., 2015, 5: 12477[2024-03-15]. . |
| 36 | DENG G, HUANG X, XIE L, et al.. Identification and expression of SAUR genes in the CAM plant Agave [J/OL]. Genes Basel., 2019, 10(7): e555[2024-03-15]. . |
| 37 | LIU Y, XIAO L, CHI J, et al.. Genome-wide identification and expression of SAUR gene family in peanut (Arachis hypogaea L.) and functional identification of AhSAUR3 in drought tolerance[J/OL]. BMC Plant Biol., 2022, 22(1): 178[2024-03-15]. . |
| [1] | 乔红梅. 红树科陆生植物竹节树的转录组分析[J]. 生物技术进展, 2024, 14(2): 228-236. |
| [2] | 王彩虹, 邵煜涵, 蒋梦媛. IL-17抑制铜绿假单胞菌感染肺上皮细胞的机制研究[J]. 生物技术进展, 2024, 14(2): 304-311. |
| [3] | 刘勤勤, 黄柏铭, 马叶子, 刘翠翠, 王洪涛, 周家喜. 急性感染条件下巨核细胞与血小板转录组变化的对比分析[J]. 生物技术进展, 2023, 13(3): 465-472. |
| [4] | 李敏敏, 夏美娟, 赵晶晶, 陈肖源, 刘翠翠, 苏培, 王洪涛, 周家喜. 人类胚胎期和成年期巨核细胞的分子特征比较[J]. 生物技术进展, 2022, 12(4): 577-583. |
| [5] | 李东翱, 刘慧泉, 王秦虎. 小麦响应禾谷镰刀菌侵染的转录组学研究进展[J]. 生物技术进展, 2021, 11(5): 610-617. |
| [6] | 柯杨, 马瑜, 朱海云, 张育辉. RNA‑seq技术在水生生物生态毒理学中的应用进展[J]. 生物技术进展, 2021, 11(4): 535-541. |
| [7] | 方汉顺,,谢永盾,曾伟伟,郭会君,熊宏春,赵林姝,古佳玉,徐延浩,刘录祥. 小麦矮秆突变体jm22d响应赤霉素处理的转录组学分析[J]. 生物技术进展, 2020, 10(5): 503-516. |
| [8] | 常文婧,赵彦艳. 激光捕获显微切割在肿瘤多组学研究中的应用进展[J]. 生物技术进展, 2020, 10(2): 130-136. |
| [9] | 黄春蒙,,朱鹏宇,王智,王晨光,杜智欣,魏霜4,张永江,付伟. 基于RNAi技术的转基因植物研究进展[J]. 生物技术进展, 2020, 10(1): 1-9. |
| [10] | 张在宝,,赵海,胡梦辉,邓丽君,王琦,李九丽,袁红雨,. 组学在花药发育研究中的应用进展Ⅰ:转录组学[J]. 生物技术进展, 2019, 9(5): 433-439. |
| [11] | 王硕,葛秀秀. 植物分枝性状研究进展[J]. 生物技术进展, 2017, 7(2): 98-101. |
| [12] | 谷培云,卢润刚,王步云,孙海,杨金利,焦雪霞,李云龙. 电生功能水对辣椒白粉病防治效果研究[J]. 生物技术进展, 2016, 6(1): 71-74. |
| [13] | 吴星波,郝俊杰,张晓艳,万述伟,李红卫,邵阳,孙吉禄. 普通菜豆抗白粉病基因SCAR标记鉴定[J]. 生物技术进展, 2013, 3(5): 357-362. |
| [14] | 吴一雷, 闫鹏程,刘充,陈禹保,赵文明. 基于高通量RNA测序数据分析的弹性云平台[J]. 生物技术进展, 2012, 2(1): 52-56. |
| [15] | 秦太辰. 作物雄性不育性在育种中的应用概评[J]. 生物技术进展, 2011, 1(2): 84-89. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||
版权所有 © 2021《生物技术进展》编辑部