


生物技术进展 ›› 2021, Vol. 11 ›› Issue (6): 783-794.DOI: 10.19586/j.2095-2341.2021.0127
收稿日期:2021-07-02
接受日期:2021-09-30
出版日期:2021-11-25
发布日期:2021-11-26
通讯作者:
彭进
作者简介:梁永弈,E-mail: ksgt00533994@163.com
基金资助:
Yongyi LIANG1( ), Yue LONG2, Jiahua XIAN1, Jun LIN1, Jin PENG2(
), Yue LONG2, Jiahua XIAN1, Jun LIN1, Jin PENG2( )
)
Received:2021-07-02
Accepted:2021-09-30
Online:2021-11-25
Published:2021-11-26
Contact:
Jin PENG
摘要:
利用可视化分析软件,客观、全面地展示和分析适体的研究现状、研究热点和发展趋势,为适体的研究提供指导和参考。以SCI数据库Web of Science平台为数据源,采用CiteSpace 5.5.R2软件对1900年1月1日至2020年12月31日的适体相关文献进行分析。共纳入文献14 939篇。在该领域发表论文最多的作者是WEIHONG TAN、KHALIL ABNOUS等;近年来突现的主题词是“wide linear range”“pg ml”“clinical diagnosis”“early diagnosis”等,是目前该领域的研究热点;作者共被引分析和文献共被引分析表明,该领域的研究主要集中在适体的体外筛选、适体构象的变化、适体在药物靶向治疗中的应用及适体生物传感器。结果表明,利用CiteSpace软件对Web of Science数据库中已发表的适体文献进行分析,揭示了该领域的研究热点从体外筛选和适体构象变化(1992—2002)到适体在药物靶向治疗和适体生物传感器中的应用(2002—2020)。结合近年来突现的主题词预测适体在药物靶向治疗和适体生物传感器中的应用仍将是未来的研究热点。
中图分类号:
梁永羿, 龙粤, 冼嘉华, 林骏, 彭进. 基于CiteSpace分析核酸适体(Aptamer)技术研究现状[J]. 生物技术进展, 2021, 11(6): 783-794.
Yongyi LIANG, Yue LONG, Jiahua XIAN, Jun LIN, Jin PENG. Analysis on the Research Status of Aptamer Technology Based on CiteSpace[J]. Current Biotechnology, 2021, 11(6): 783-794.
| 排名 | 发文作者 | 国家 | 发文量/篇 | 发文起始年份 | 占总文献比例/% |
|---|---|---|---|---|---|
| 1 | WEIHONG TAN | 美国 | 241 | 2006 | 1.613 |
| 2 | KHALIL ABNOUS | 伊朗 | 125 | 2015 | 0.837 |
| 3 | KEMIN WANG | 中国 | 119 | 2008 | 0.797 |
| 4 | MOHAMMAD RAMEZANI | 伊朗 | 116 | 2015 | 0.776 |
| 5 | RUO YUAN | 中国 | 115 | 2010 | 0.770 |
| 6 | SEYED MOHAMMAD TAGHDISI | 伊朗 | 107 | 2015 | 0.716 |
| 7 | ZHOUPING WANG | 中国 | 95 | 2013 | 0.636 |
| 8 | NUO DUAN | 中国 | 72 | 2013 | 0.482 |
| 9 | YAQIN CHAI | 中国 | 72 | 2010 | 0.482 |
| 10 | SHIJIA WU | 中国 | 68 | 2013 | 0.455 |
表1 发文量前10作者(1992—2020年)
Table 1 Top ten authors in the publications(1992—2020)
| 排名 | 发文作者 | 国家 | 发文量/篇 | 发文起始年份 | 占总文献比例/% |
|---|---|---|---|---|---|
| 1 | WEIHONG TAN | 美国 | 241 | 2006 | 1.613 |
| 2 | KHALIL ABNOUS | 伊朗 | 125 | 2015 | 0.837 |
| 3 | KEMIN WANG | 中国 | 119 | 2008 | 0.797 |
| 4 | MOHAMMAD RAMEZANI | 伊朗 | 116 | 2015 | 0.776 |
| 5 | RUO YUAN | 中国 | 115 | 2010 | 0.770 |
| 6 | SEYED MOHAMMAD TAGHDISI | 伊朗 | 107 | 2015 | 0.716 |
| 7 | ZHOUPING WANG | 中国 | 95 | 2013 | 0.636 |
| 8 | NUO DUAN | 中国 | 72 | 2013 | 0.482 |
| 9 | YAQIN CHAI | 中国 | 72 | 2010 | 0.482 |
| 10 | SHIJIA WU | 中国 | 68 | 2013 | 0.455 |
| 聚类 ID | 大小 | 轮廓值 | 平均引用年限 | 代表作者 | 研究内容 |
|---|---|---|---|---|---|
| 0 | 40 | 0.991 | 2010 | WEIHONG TAN | 纳米材料与适体结合用于肿瘤细胞的鉴别、分析 |
| 1 | 28 | 0.972 | 2015 | KEMIN WANG | 适体用于生物分子的检测 |
| 2 | 19 | 0.979 | 2013 | ZHILIANG JIANG,KUN WANG | 适体结合共振散射光谱检测靶物质 |
| 3 | 19 | 0.998 | 2015 | MOHAMMAD RAMEZANI,ZHOUPING WANG等 | 适体用于药物的靶向治疗及生物传感器的研究 |
表2 文献作者聚类信息(1992—2020年)
Table 2 Clusters information of the authors(1992—2020)
| 聚类 ID | 大小 | 轮廓值 | 平均引用年限 | 代表作者 | 研究内容 |
|---|---|---|---|---|---|
| 0 | 40 | 0.991 | 2010 | WEIHONG TAN | 纳米材料与适体结合用于肿瘤细胞的鉴别、分析 |
| 1 | 28 | 0.972 | 2015 | KEMIN WANG | 适体用于生物分子的检测 |
| 2 | 19 | 0.979 | 2013 | ZHILIANG JIANG,KUN WANG | 适体结合共振散射光谱检测靶物质 |
| 3 | 19 | 0.998 | 2015 | MOHAMMAD RAMEZANI,ZHOUPING WANG等 | 适体用于药物的靶向治疗及生物传感器的研究 |
| 频率 | 主题词 | 最早 出现年 | 聚类 | 突现值 | 主题词 | 最早 出现年 | 聚类 | 中心性 | 主题词 | 最早 出现年 | 聚类 |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 2 398 | Detection limits | 2002 | 1 | 121.75 | Vitro selections | 1992 | 0 | 0.21 | Conformational change | 1994 | 5 |
| 1 122 | DNA aptamers | 1994 | 0 | 89.99 | RNA aptamers | 1996 | 0 | 0.13 | DNA aptamers | 1994 | 0 |
| 961 | Gold nanoparticles | 2006 | 1 | 51.98 | Secondary structure | 1994 | 0 | 0.12 | High affinities | 1994 | 0 |
| 958 | High selectivity | 2004 | 1 | 42.82 | Pg ml | 2018 | 1 | 0.11 | Inhibitory effects | 1996 | 6 |
| 953 | High affinities | 1994 | 0 | 42.78 | Wide linear range | 2018 | 1 | 0.09 | High specificity | 2000 | 1 |
表3 高影响力主题词(1992—2020年)
Table 3 High impact terms(1992—2020)
| 频率 | 主题词 | 最早 出现年 | 聚类 | 突现值 | 主题词 | 最早 出现年 | 聚类 | 中心性 | 主题词 | 最早 出现年 | 聚类 |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 2 398 | Detection limits | 2002 | 1 | 121.75 | Vitro selections | 1992 | 0 | 0.21 | Conformational change | 1994 | 5 |
| 1 122 | DNA aptamers | 1994 | 0 | 89.99 | RNA aptamers | 1996 | 0 | 0.13 | DNA aptamers | 1994 | 0 |
| 961 | Gold nanoparticles | 2006 | 1 | 51.98 | Secondary structure | 1994 | 0 | 0.12 | High affinities | 1994 | 0 |
| 958 | High selectivity | 2004 | 1 | 42.82 | Pg ml | 2018 | 1 | 0.11 | Inhibitory effects | 1996 | 6 |
| 953 | High affinities | 1994 | 0 | 42.78 | Wide linear range | 2018 | 1 | 0.09 | High specificity | 2000 | 1 |
| 聚类 ID | 大小 | 轮廓值 | 平均引用年限 | 主要主题词 |
|---|---|---|---|---|
| 0 | 90 | 0.612 | 1999 | High affinities, SELEX, vitro selections, target molecules, secondary structure |
| 1 | 69 | 0.784 | 2008 | Detection limits, gold nanoparticles, high selectivity, sensitive detection, high sensitivity, high specificity, wide linear range, early diagnosis |
| 2 | 54 | 0.784 | 1996 | Complex formation, solution structure, RNA structure, structural motifs, structural basis |
| 3 | 37 | 0.824 | 1997 | Human thrombin, circular dichroism, vascular endothelial growth factor |
| 4 | 32 | 0.735 | 1998 | Model system, quadruplex structure, three‑dimensional structure, specific inhibitors |
| 5 | 27 | 0.888 | 1997 | Conformational change, single‑stranded DNA, tertiary structure, double‑stranded DNA, allosteric ribozymes, catalytic activity |
| 6 | 10 | 0.998 | 1996 | Inhibitory effects, antiproliferative effects, systematic approach |
表4 主题词聚类信息(1992—2020年)
Table 4 Clusters information of the terms(1992—2020)
| 聚类 ID | 大小 | 轮廓值 | 平均引用年限 | 主要主题词 |
|---|---|---|---|---|
| 0 | 90 | 0.612 | 1999 | High affinities, SELEX, vitro selections, target molecules, secondary structure |
| 1 | 69 | 0.784 | 2008 | Detection limits, gold nanoparticles, high selectivity, sensitive detection, high sensitivity, high specificity, wide linear range, early diagnosis |
| 2 | 54 | 0.784 | 1996 | Complex formation, solution structure, RNA structure, structural motifs, structural basis |
| 3 | 37 | 0.824 | 1997 | Human thrombin, circular dichroism, vascular endothelial growth factor |
| 4 | 32 | 0.735 | 1998 | Model system, quadruplex structure, three‑dimensional structure, specific inhibitors |
| 5 | 27 | 0.888 | 1997 | Conformational change, single‑stranded DNA, tertiary structure, double‑stranded DNA, allosteric ribozymes, catalytic activity |
| 6 | 10 | 0.998 | 1996 | Inhibitory effects, antiproliferative effects, systematic approach |
| 聚类 ID | 大小 | 轮廓值 | 平均引用年限 | 主要被引作者 |
|---|---|---|---|---|
| 0 | 59 | 0.871 | 2011 | LIU J W, WANG J, WANG Y, ZHANG Y, LI H, LI J |
| 1 | 58 | 0.841 | 2004 | ELLINGTON A D, TUERK C, BOCK L C, ZHOU J H, TOMBELLI S, KIM Y S |
| 2 | 49 | 0.911 | 1998 | HUIZENGA D E, FAMULOK M, WILSON D S, JENISON R D |
| 3 | 27 | 0.967 | 1994 | MACAYA R F, WANG K Y, PADMANABHAN K |
| 4 | 18 | 1.000 | 1993 | KARN J, HEAPHY S, SULLENGER B A |
| 5 | 13 | 0.938 | 1995 | ROBERTSON D L, MILLIGAN J F, PUGLISI J D, HEUS H A |
| 6 | 10 | 0.991 | 1993 | PABORSKY L R, WU Q Y, GRIFFIN L C |
表5 作者共被引聚类信息(1992—2020年)
Table 5 Clusters information of cited authors(1992—2020)
| 聚类 ID | 大小 | 轮廓值 | 平均引用年限 | 主要被引作者 |
|---|---|---|---|---|
| 0 | 59 | 0.871 | 2011 | LIU J W, WANG J, WANG Y, ZHANG Y, LI H, LI J |
| 1 | 58 | 0.841 | 2004 | ELLINGTON A D, TUERK C, BOCK L C, ZHOU J H, TOMBELLI S, KIM Y S |
| 2 | 49 | 0.911 | 1998 | HUIZENGA D E, FAMULOK M, WILSON D S, JENISON R D |
| 3 | 27 | 0.967 | 1994 | MACAYA R F, WANG K Y, PADMANABHAN K |
| 4 | 18 | 1.000 | 1993 | KARN J, HEAPHY S, SULLENGER B A |
| 5 | 13 | 0.938 | 1995 | ROBERTSON D L, MILLIGAN J F, PUGLISI J D, HEUS H A |
| 6 | 10 | 0.991 | 1993 | PABORSKY L R, WU Q Y, GRIFFIN L C |
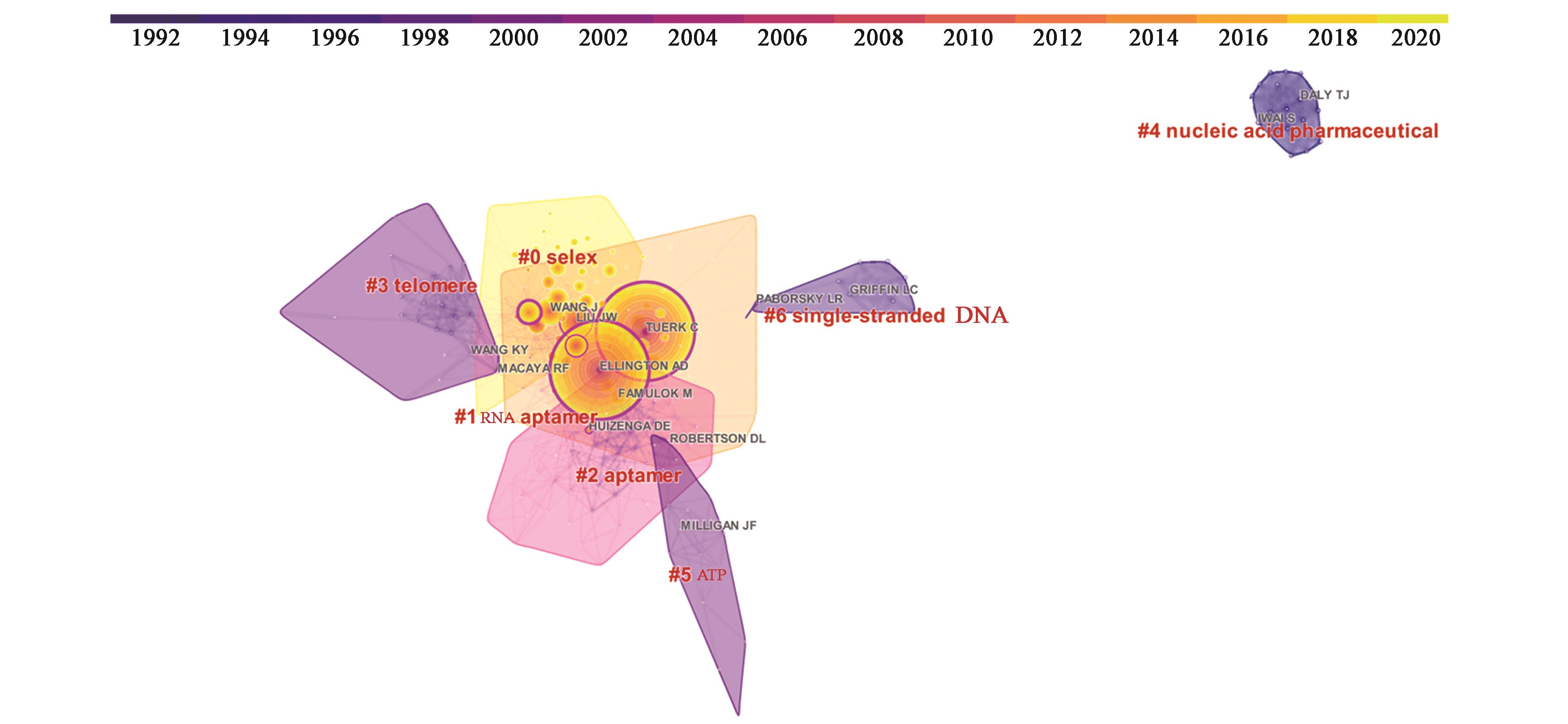
图3 作者共被引聚类图谱(1992—2020年)注:Citespace 设置时间切片为 2,每个切片 TOP N=43,剪切方式无,得到 254个节点、1 403条连线的作者共被引图谱;聚类标签为关键词,聚类算法为LLR,Q值>0.3、平均轮廓值s>0.5
Fig.3 Cluster view of cited authors(1992—2020)
| 被引频率 | 作者 | 最早 发文年 | 聚类 | 突现值 | 作者 | 最早 发文年 | 聚类 | 中心性 | 作者 | 最早 发文年 | 聚类 |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 3 561 | ELLINGTON A D | 1992 | 1 | 124.68 | ZHOU J H | 2018 | 1 | 0.30 | WANG Y | 1993 | 0 |
| 3 484 | TUERK C | 1992 | 1 | 120.13 | ZHANG Y | 2016 | 0 | 0.29 | ELLINGTON A D | 1992 | 1 |
| 1 244 | LIU J W | 2006 | 0 | 111.25 | LI H | 2016 | 0 | 0.26 | TUERK C | 1992 | 1 |
| 1 168 | WANG J | 2006 | 0 | 104.05 | LI J | 2015 | 0 | 0.18 | BOCK L C | 1993 | 1 |
| 1 101 | BOCK L C | 1993 | 1 | 102.04 | PAVLOV V | 2006 | 0 | 0.12 | HUIZENGA D E | 1996 | 2 |
| 1 055 | WANG Y | 1993 | 0 | 99.75 | LI X | 2016 | 0 | 0.12 | PABORSKY L R | 1994 | 6 |
表6 高影响被引作者(1992—2020年)
Table 6 High impact cited authors(1992—2020)
| 被引频率 | 作者 | 最早 发文年 | 聚类 | 突现值 | 作者 | 最早 发文年 | 聚类 | 中心性 | 作者 | 最早 发文年 | 聚类 |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 3 561 | ELLINGTON A D | 1992 | 1 | 124.68 | ZHOU J H | 2018 | 1 | 0.30 | WANG Y | 1993 | 0 |
| 3 484 | TUERK C | 1992 | 1 | 120.13 | ZHANG Y | 2016 | 0 | 0.29 | ELLINGTON A D | 1992 | 1 |
| 1 244 | LIU J W | 2006 | 0 | 111.25 | LI H | 2016 | 0 | 0.26 | TUERK C | 1992 | 1 |
| 1 168 | WANG J | 2006 | 0 | 104.05 | LI J | 2015 | 0 | 0.18 | BOCK L C | 1993 | 1 |
| 1 101 | BOCK L C | 1993 | 1 | 102.04 | PAVLOV V | 2006 | 0 | 0.12 | HUIZENGA D E | 1996 | 2 |
| 1 055 | WANG Y | 1993 | 0 | 99.75 | LI X | 2016 | 0 | 0.12 | PABORSKY L R | 1994 | 6 |
| 聚类 ID | 大小 | 轮廓值 | 平均引用年限 | 主要研究内容 | 主要文献 |
|---|---|---|---|---|---|
| 0 | 56 | 0.867 | 1994 | 体外筛选适体技术研究 | [ |
| 1 | 42 | 0.822 | 2006 | 适体在生物传感器方面的研究 | [ |
| 2 | 40 | 0.785 | 2010 | 适体在诊断治疗上的应用研究 | [ |
| 3 | 38 | 0.887 | 2015 | 适体在药物靶向传送方面的研究 | [ |
| 4 | 27 | 0.885 | 2000 | 开发基于适体的通用荧光传感器 | [ |
| 5 | 23 | 0.821 | 1997 | 开发特异性识别细胞的适体 | [ |
| 6 | 23 | 0.979 | 1991 | 开发基于适体的凝血酶抑制剂 | [ |
| 7 | 6 | 0.988 | 2003 | 适体在参与调节生物体代谢过程的应用 | [ |
表7 文献共被引聚类信息(1992—2020年)
Table 7 Clusters information of cited documents(1992—2020)
| 聚类 ID | 大小 | 轮廓值 | 平均引用年限 | 主要研究内容 | 主要文献 |
|---|---|---|---|---|---|
| 0 | 56 | 0.867 | 1994 | 体外筛选适体技术研究 | [ |
| 1 | 42 | 0.822 | 2006 | 适体在生物传感器方面的研究 | [ |
| 2 | 40 | 0.785 | 2010 | 适体在诊断治疗上的应用研究 | [ |
| 3 | 38 | 0.887 | 2015 | 适体在药物靶向传送方面的研究 | [ |
| 4 | 27 | 0.885 | 2000 | 开发基于适体的通用荧光传感器 | [ |
| 5 | 23 | 0.821 | 1997 | 开发特异性识别细胞的适体 | [ |
| 6 | 23 | 0.979 | 1991 | 开发基于适体的凝血酶抑制剂 | [ |
| 7 | 6 | 0.988 | 2003 | 适体在参与调节生物体代谢过程的应用 | [ |
| 共被引频率 | 作者(国家) | 聚类 | 发表年份 | 主要内容 | 文献 |
|---|---|---|---|---|---|
| 504 | KEEFE A D(美国) | 2 | 2010 | 适体药物应用方面进展 | [ |
| 438 | LIU J W(美国) | 2 | 2009 | 适体在生物传感器方面的进展 | [ |
| 292 | SHANGGUAN D(美国) | 2 | 2006 | 筛选特异性识别白血病细胞的适体 | [ |
| 279 | WILLNER I(以色列) | 1 | 2010 | 电子适体传感器应用进展 | [ |
| 260 | CHO E J(美国) | 2 | 2009 | 电子适体传感器应用进展 | [ |
| 253 | LIU J W(美国) | 1 | 2005 | 适体与纳米金粒子结合用于生物传感器,靶标与适体结合造成连接在适体上的纳米金释放出来,从而产生颜色变化;可作为一种通用的传感器设计 | [ |
| 252 | FANG X H(中国) | 2 | 2010 | 适体用于特异性识别肿瘤细胞 | [ |
| 248 | SONG S P(中国) | 1 | 2008 | 适体在生物传感器方面的进展 | [ |
| 247 | ILIUK A B(美国) | 2 | 2011 | 适体在生物分析中的应用进展 | [ |
| 247 | TAN W H(中国/美国) | 2 | 2013 | 基于细胞选择的适体在生物分析中的应用进展 | [ |
| 247 | FAMULOK M(德国) | 2 | 2007 | 功能适体和适体酶在诊断和治疗上的应用进展 | [ |
表8 文献共被引频次排名(1992—2020年)
Table 8 Ranking of total citation frequency of documents(1992—2020)
| 共被引频率 | 作者(国家) | 聚类 | 发表年份 | 主要内容 | 文献 |
|---|---|---|---|---|---|
| 504 | KEEFE A D(美国) | 2 | 2010 | 适体药物应用方面进展 | [ |
| 438 | LIU J W(美国) | 2 | 2009 | 适体在生物传感器方面的进展 | [ |
| 292 | SHANGGUAN D(美国) | 2 | 2006 | 筛选特异性识别白血病细胞的适体 | [ |
| 279 | WILLNER I(以色列) | 1 | 2010 | 电子适体传感器应用进展 | [ |
| 260 | CHO E J(美国) | 2 | 2009 | 电子适体传感器应用进展 | [ |
| 253 | LIU J W(美国) | 1 | 2005 | 适体与纳米金粒子结合用于生物传感器,靶标与适体结合造成连接在适体上的纳米金释放出来,从而产生颜色变化;可作为一种通用的传感器设计 | [ |
| 252 | FANG X H(中国) | 2 | 2010 | 适体用于特异性识别肿瘤细胞 | [ |
| 248 | SONG S P(中国) | 1 | 2008 | 适体在生物传感器方面的进展 | [ |
| 247 | ILIUK A B(美国) | 2 | 2011 | 适体在生物分析中的应用进展 | [ |
| 247 | TAN W H(中国/美国) | 2 | 2013 | 基于细胞选择的适体在生物分析中的应用进展 | [ |
| 247 | FAMULOK M(德国) | 2 | 2007 | 功能适体和适体酶在诊断和治疗上的应用进展 | [ |
| 中心性 | 作者(国家) | 聚类 | 发表年份 | 主要内容 | 文献 |
|---|---|---|---|---|---|
| 0.55 | NUTIU R(加拿大) | 1 | 2003 | 设计了一种基于适体的荧光检测系统,含荧光基团的单链核酸与含淬灭基团的单链核酸在没有靶标存在时均与适体互补形成双链,荧光基团与含淬灭基团接近无法发出荧光,当存在靶标时,适体与靶标结合,含淬灭基团的单链核酸被释放,从而发出荧光 | [ |
| 0.41 | WILSON D S(美国) | 0 | 1999 | 功能性核酸体外筛选方法的进展 | [ |
| 0.34 | HUIZENGA D E(美国) | 0 | 1995 | 利用体外筛选的方法,分离出结合腺苷/ATP的适体;并提出了一个ATP结合DNA的模型结构 | [ |
| 0.29 | LIU J W(美国) | 2 | 2009 | 适体在生物传感器方面的应用进展 | [ |
| 0.29 | BOCK L C(美国) | 6 | 1992 | 筛选特异结合并抑制凝血酶活性的单链DNA适体 | [ |
| 0.18 | JHAVERI S(美国) | 4 | 2000 | 建立通用的筛选有信号转导功能的适体方法(有荧光标记,且适体与靶标结合构象发生变化时荧光信号强度会发生变化);在适体筛选的序列中引入少量荧光标记的尿苷,获得的腺苷适体在腺苷同源物ATP存在下,其荧光会增强,通过观察荧光强度变化即可确认靶标物质的存在 | [ |
| 0.17 | LIU J W(美国) | 1 | 2005 | 适体与纳米金粒子结合用于生物传感器,靶标与适体结合造成连接在适体上的纳米金释放出来,从而产生颜色变化;可作为一种通用的传感器设计 | [ |
| 0.15 | GOLD L(美国) | 5 | 1995 | SELEX技术进展 | [ |
| 0.14 | ILIUK A B(美国) | 2 | 2011 | 适体在生物分析中的应用进展 | [ |
| 0.13 | SHANGGUAN D(美国) | 2 | 2006 | 特异性识别白血病细胞的适体 | [ |
| 0.12 | DANIELS D A(美国) | 5 | 2003 | 利用SELEX技术筛选特异性识别肿瘤细胞的适体 | [ |
| 0.11 | STOJANOVIC M N (美国) | 4 | 2001 | 开发基于适体的可卡因荧光传感器。适体5′端和3′端分别有荧光基团和淬灭基团,无靶标时,荧光基团和淬灭基团分离,可以发出荧光;当可卡因存在时,适体结合可卡因并发生构象变化,荧光基团和淬灭基团靠近,荧光被淬灭 | [ |
| 0.11 | SASSANFAR M(美国) | 0 | 1993 | 开发了一种用于体外筛选ATP适体的新方法 | [ |
| 0.11 | MORRIS K N(美国) | 5 | 1998 | 利用SELEX技术筛选特异性识别人红细胞膜的适体 | [ |
| 0.10 | WILLNER I(以色列) | 1 | 2010 | 电子适体传感器应用进展 | [ |
| 0.10 | DENG B(加拿大) | 3 | 2014 | 蛋白质适体研究进展 | [ |
| 0.10 | JIANG F(美国) | 0 | 1996 | 研究RNA适体的空间结构 | [ |
表9 文献共被引中心性值排名(1992—2020年)
Table 9 Centrality ranking of total citation documents(1992—2020)
| 中心性 | 作者(国家) | 聚类 | 发表年份 | 主要内容 | 文献 |
|---|---|---|---|---|---|
| 0.55 | NUTIU R(加拿大) | 1 | 2003 | 设计了一种基于适体的荧光检测系统,含荧光基团的单链核酸与含淬灭基团的单链核酸在没有靶标存在时均与适体互补形成双链,荧光基团与含淬灭基团接近无法发出荧光,当存在靶标时,适体与靶标结合,含淬灭基团的单链核酸被释放,从而发出荧光 | [ |
| 0.41 | WILSON D S(美国) | 0 | 1999 | 功能性核酸体外筛选方法的进展 | [ |
| 0.34 | HUIZENGA D E(美国) | 0 | 1995 | 利用体外筛选的方法,分离出结合腺苷/ATP的适体;并提出了一个ATP结合DNA的模型结构 | [ |
| 0.29 | LIU J W(美国) | 2 | 2009 | 适体在生物传感器方面的应用进展 | [ |
| 0.29 | BOCK L C(美国) | 6 | 1992 | 筛选特异结合并抑制凝血酶活性的单链DNA适体 | [ |
| 0.18 | JHAVERI S(美国) | 4 | 2000 | 建立通用的筛选有信号转导功能的适体方法(有荧光标记,且适体与靶标结合构象发生变化时荧光信号强度会发生变化);在适体筛选的序列中引入少量荧光标记的尿苷,获得的腺苷适体在腺苷同源物ATP存在下,其荧光会增强,通过观察荧光强度变化即可确认靶标物质的存在 | [ |
| 0.17 | LIU J W(美国) | 1 | 2005 | 适体与纳米金粒子结合用于生物传感器,靶标与适体结合造成连接在适体上的纳米金释放出来,从而产生颜色变化;可作为一种通用的传感器设计 | [ |
| 0.15 | GOLD L(美国) | 5 | 1995 | SELEX技术进展 | [ |
| 0.14 | ILIUK A B(美国) | 2 | 2011 | 适体在生物分析中的应用进展 | [ |
| 0.13 | SHANGGUAN D(美国) | 2 | 2006 | 特异性识别白血病细胞的适体 | [ |
| 0.12 | DANIELS D A(美国) | 5 | 2003 | 利用SELEX技术筛选特异性识别肿瘤细胞的适体 | [ |
| 0.11 | STOJANOVIC M N (美国) | 4 | 2001 | 开发基于适体的可卡因荧光传感器。适体5′端和3′端分别有荧光基团和淬灭基团,无靶标时,荧光基团和淬灭基团分离,可以发出荧光;当可卡因存在时,适体结合可卡因并发生构象变化,荧光基团和淬灭基团靠近,荧光被淬灭 | [ |
| 0.11 | SASSANFAR M(美国) | 0 | 1993 | 开发了一种用于体外筛选ATP适体的新方法 | [ |
| 0.11 | MORRIS K N(美国) | 5 | 1998 | 利用SELEX技术筛选特异性识别人红细胞膜的适体 | [ |
| 0.10 | WILLNER I(以色列) | 1 | 2010 | 电子适体传感器应用进展 | [ |
| 0.10 | DENG B(加拿大) | 3 | 2014 | 蛋白质适体研究进展 | [ |
| 0.10 | JIANG F(美国) | 0 | 1996 | 研究RNA适体的空间结构 | [ |
| 突现值 | 作者(国家) | 聚类 | 发表年份 | 主要内容 | 文献 |
|---|---|---|---|---|---|
| 144.29 | KEEFE A D(美国) | 2 | 2010 | 适体药物应用方面的进展 | [ |
| 107.59 | ZHOU J H(美国) | 3 | 2017 | 适体作为药物靶向传送剂在临床转化上存在的挑战 | [ |
| 103.52 | LIU J W(美国) | 2 | 2009 | 适体在生物传感器方面的应用 | [ |
| 87.29 | ILIUK A B(美国) | 2 | 2011 | 适体在生物分析中的应用 | [ |
| 81.23 | SHANGGUAN D(美国) | 2 | 2006 | 特异性识别白血病细胞的适体 | [ |
表10 文献共被引突现值排名(1992—2020年)
Table 10 Bursts ranking of total citation documents(1992—2020)
| 突现值 | 作者(国家) | 聚类 | 发表年份 | 主要内容 | 文献 |
|---|---|---|---|---|---|
| 144.29 | KEEFE A D(美国) | 2 | 2010 | 适体药物应用方面的进展 | [ |
| 107.59 | ZHOU J H(美国) | 3 | 2017 | 适体作为药物靶向传送剂在临床转化上存在的挑战 | [ |
| 103.52 | LIU J W(美国) | 2 | 2009 | 适体在生物传感器方面的应用 | [ |
| 87.29 | ILIUK A B(美国) | 2 | 2011 | 适体在生物分析中的应用 | [ |
| 81.23 | SHANGGUAN D(美国) | 2 | 2006 | 特异性识别白血病细胞的适体 | [ |
| 1 | ELLINGTON A D, SZOSTAK J W. In vitro selection of RNA molecules that bind specific ligands[J]. Nature, 1990, 346(6287):818-822. |
| 2 | TUERK C, GOLD L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase[J]. Science, 1990, 249(4968):505-510. |
| 3 | CHEN C, HU Z, LIU S, et al.. Emerging trends in regenerative medicine: a scientometric analysis in CiteSpace[J]. Expert. Opin. Biol. Ther., 2012, 12(5):593-608. |
| 4 | WANG S, ZHANG L, WAN S, et al.. Aptasensor with expanded nucleotide using DNA nanotetrahedra for electrochemical detection of cancerous exosomes[J]. ACS Nano, 2017, 11(4):3943-3949. |
| 5 | JIANG Y, SHI M, LIU Y, et al.. Aptamer/AuNP biosensor for colorimetric profiling of exosomal proteins[J]. Angew. Chem. Int. Ed. Engl., 2017, 56(39):11916-11920. |
| 6 | LIU C, ZHAO J, TIAN F, et al.. Low-cost thermophoretic profiling of extracellular-vesicle surface proteins for the early detection and classification of cancers[J]. Nat. Biomed. Eng., 2019, 3(3):183-193. |
| 7 | RAMEZANI M, MOHAMMAD D N, LAVAEE P, et al.. A novel colorimetric triple-helix molecular switch aptasensor for ultrasensitive detection of tetracycline[J]. Biosens. Bioelectron., 2015, 70:181-187. |
| 8 | EMRANI A S, DANESH N M, LAVAEE P, et al.. Colorimetric and fluorescence quenching aptasensors for detection of streptomycin in blood serum and milk based on double-stranded DNA and gold nanoparticles[J]. Food Chem., 2016, 190:115-121. |
| 9 | MOHAMMAD D N, RAMEZANI M, SARRESHTEHDAR E A, et al.. A novel electrochemical aptasensor based on arch-shape structure of aptamer-complimentary strand conjugate and exonuclease I for sensitive detection of streptomycin[J]. Biosens. Bioelectron., 2016, 75:123-128. |
| 10 | RAMEZANI M, DANESH N M, LAVAEE P, et al.. A selective and sensitive fluorescent aptasensor for detection of kanamycin based on catalytic recycling activity of exonuclease III and gold nanoparticles[J]. Sens. Actuat. B Chem., 2016, 222:1-7. |
| 11 | ALIBOLANDI M, MOHAMMADI M, TAGHDISI S M, et al.. Fabrication of aptamer decorated dextran coated nano-graphene oxide for targeted drug delivery[J]. Carbohydr. Polym., 2017, 155:218-229. |
| 12 | TAGHAVI S, RAMEZANI M, ALIBOLANDI M, et al.. Chitosan-modified PLGA nanoparticles tagged with 5TR1 aptamer for in vivo tumor-targeted drug delivery[J]. Cancer Lett., 2017, 400:1-8. |
| 13 | LI S, ZHENG Y, ZOU Q, et al.. Engineering and application of a myoglobin binding split aptamer[J]. Anal. Chem., 2020, 92(21):14576-14581. |
| 14 | TANG J, LEI Y, HE X, et al.. Recognition-driven remodeling of dual-split aptamer triggering in situ hybridization chain reaction for activatable and autonomous identification of cancer cells[J]. Anal. Chem., 2020, 92(15):10839-10846. |
| 15 | LI S, ZHENG Y, LIU Y, et al.. Investigation of the interaction between split aptamer and vascular endothelial growth factor 165 using single molecule force spectroscopy[J]. J. Mol. Recognit., 2020, 33(5):1-6. |
| 16 | YUAN B, SUN Y, GUO Q, et al.. High signal-to-background ratio detection of cancer cells with activatable strategy based on target-induced self-assembly of split aptamers[J]. Anal. Chem., 2017, 89(17):9347-9353. |
| 17 | LEI Y, TANG J, SHI H, et al.. Nature-inspired smart DNA nanodoctor for activatable in vivo cancer imaging and in situ drug release based on recognition-triggered assembly of split aptamer[J]. Anal. Chem., 2016, 88(23):11699-11706. |
| 18 | STOJANOVIC M N, PRADA P D, LANDRY D W. Fluorescent sensors based on aptamer self-assembly[J]. J. Am. Chem. Soc., 2000, 122(46):11547-11548. |
| 19 | MA X, GUO Z, MAO Z, et al.. Colorimetric theophylline aggregation assay using an RNA aptamer and non-crosslinking gold nanoparticles[J]. Microchim. Acta, 2018, 185(1):1-7. |
| 20 | LIU X, SHI L, HUA X, et al.. Target-induced conjunction of split aptamer fragments and assembly with a water-soluble conjugated polymer for improved protein detection[J]. Acs. Appl. Mater. Interfaces., 2014, 6(5):3406-3412. |
| 21 | LIU X, YANG Y, HUA X, et al.. An improved turn-on aptasensor for thrombin detection using split aptamer fragments and graphene oxide[J]. Chin. J. Chem., 2015, 33(8):981-986. |
| 22 | LUO Y, YU H, ALKHAMIS O, et al.. Label-free, visual detection of small molecules using highly target-responsive multi-module split aptamer constructs[J]. Anal. Chem., 2019, 91(11):7199-7207. |
| 23 | ABNOUS K, DANESH N M, RAMEZANI M, et al.. A novel amplified double-quenching aptasensor for cocaine detection based on split aptamer and silica nanoparticles[J]. Anal. Methods, 2018, 10(26):3232-3236. |
| 24 | YANG J, DOU B, YUAN R, et al.. Proximity binding and metal ion-dependent DNAzyme cyclic amplification-integrated aptasensor for label-free and sensitive electrochemical detection of thrombin[J]. Anal. Chem., 2016, 88(16):8218-8223. |
| 25 | JIANG X, WANG H, WANG H, et al.. Electrochemiluminescence biosensor based on 3-D DNA nanomachine signal probe powered by protein-aptamer binding complex for ultrasensitive Mucin 1 detection[J]. Anal. Chem., 2017, 89(7):4280-4286. |
| 26 | YANG J, DOU B, YUAN R, et al.. Aptamer/protein proximity binding-triggered molecular machine for amplified electrochemical sensing of thrombin[J]. Anal. Chem., 2017, 89(9):5138-5143. |
| 27 | DA H, LIU H, ZHENG Y, et al.. A highly sensitive VEGF165 photoelectrochemical biosensor fabricated by assembly of aptamer bridged DNA networks[J]. Biosens. Bioelectron., 2018, 101:213-218. |
| 28 | NIE Y, YUAN X, ZHANG P, et al.. Versatile and ultrasensitive electrochemiluminescence biosensor for biomarker detection based on nonenzymatic amplification and aptamer-triggered emitter release[J]. Anal. Chem., 2019, 91(5):3452-3458. |
| 29 | WILSON D S, SZOSTAK J W. In vitro selection of functional nucleic acids[J]. Annu. Rev. Biochem., 1999, 68(1):611-647. |
| 30 | HUIZENGA D E, SZOSTAK J W. A DNA aptamer that binds adenosine and ATP[J]. Biochemistry, 1995, 34(2):656-665. |
| 31 | SASSANFAR M, SZOSTAK J W. An RNA motif that binds ATP[J]. Nature, 1993, 364(6437):550-553. |
| 32 | JIANG F, KUMAR R A, JONES R A, et al.. Structural basis of RNA folding and recognition in an AMP-RNA aptamer complex[J]. Nature, 1996, 382(6587):183-186. |
| 33 | WILLNER I, ZAYATS M. Electronic aptamer-based sensors[J]. Angew. Chem. Int. Ed. Engl., 2007, 46(34):6408-6418. |
| 34 | LIU J, LU Y. Fast colorimetric sensing of adenosine and cocaine based on a general sensor design involving aptamers and nanoparticles[J]. Angew. Chem. Int. Ed. Engl., 2005, 45(1):90-94. |
| 35 | SONG S, WANG L, LI J, et al.. Aptamer-based biosensors[J]. Trends Anal. Chem., 2008, 27(2):108-117. |
| 36 | NUTIU R, LI Y. Structure-switching signaling aptamers[J]. J. Am. Chem. Soc., 2003, 125(16):4771-4778. |
| 37 | SHANGGUAN D, LI Y, TANG Z, et al.. Aptamers evolved from live cells as effective molecular probes for cancer study[J]. Proc. Natl. Acad. Sci. USA, 2006, 103(32):11838-11843. |
| 38 | TAN W, DONOVAN M J, JIANG J. Aptamers from cell-based selection for bioanalytical applications[J]. Chem. Rev., 2013, 113(4):2842-2862. |
| 39 | KEEFE A D, PAI S, ELLINGTON A. Aptamers as therapeutics[J]. Nat. Rev. Drug Discov., 2010, 9(7):537-550. |
| 40 | LIU J, CAO Z, LU Y. Functional nucleic acid sensors[J]. Chem. Rev., 2009, 109(5):1948-1998. |
| 41 | CHO E J, LEE J W, ELLINGTON A D. Applications of aptamers as sensors[J]. Annu. Rev. Anal. Chem., 2009, 2:241-264. |
| 42 | FANG X, TAN W. Aptamers generated from cell-SELEX for molecular medicine: a chemical biology approach[J]. Acc. Chem. Res., 2010, 43(1):48-57. |
| 43 | ILIUK A B, HU L, TAO W A. Aptamer in bioanalytical applications[J]. Anal. Chem., 2011, 83(12):4440-4452. |
| 44 | FAMULOK M, HARTIG J S, MAYER G. Functional aptamers and aptazymes in biotechnology, diagnostics, and therapy[J]. Chem. Rev., 2007, 107(9):3715-3743. |
| 45 | DENG B, LIN Y, WANG C, et al.. Aptamer binding assays for proteins: the thrombin example——a review[J]. Anal. Chim. Acta, 2014, 837:1-15. |
| 46 | ZHOU J, ROSSI J. Aptamers as targeted therapeutics: current potential and challenges[J]. Nat. Rev. Drug Discov., 2017, 16(3):181-202. |
| 47 | JHAVERI S, RAJENDRAN M, ELLINGTON A D. In vitro selection of signaling aptamers[J]. Nat. Biotechnol., 2000, 18(12):1293-1297. |
| 48 | STOJANOVIC M N, DE PRADA P, LANDRY D W. Aptamer-based folding fluorescent sensor for cocaine[J]. J. Am. Chem. Soc., 2001, 123(21):4928-4931. |
| 49 | DANIELS D A, CHEN H, HICKE B J, et al.. A tenascin-C aptamer identified by tumor cell SELEX: systematic evolution of ligands by exponential enrichment[J]. Proc. Natl. Acad. Sci. USA, 2003, 100(26):15416-15421. |
| 50 | GOLD L, POLISKY B, UHLENBECK O, et al.. Diversity of oligonucleotide functions[J]. Annu. Rev. Biochem., 1995, 64(1):763-797. |
| 51 | MORRIS K N, JENSEN K B, JULIN C M, et al.. High affinity ligands from in vitro selection: complex targets[J]. Proc. Natl. Acad. Sci. USA, 1998, 95(6):2902-2927. |
| 52 | BOCK L C, GRIFFIN L C, LATHAM J A, et al.. Selection of single-stranded DNA molecules that bind and inhibit human thrombin[J]. Nature, 1992, 355(6360):564-566. |
| 53 | MACAYA R F, SCHULTZE P, SMITH F W, et al.. Thrombin-binding DNA aptamer forms a unimolecular quadruplex structure in solution[J]. Proc. Natl. Acad. Sci. USA, 1993, 90(8):3745-3749. |
| 54 | MANDAL M, BREAKER R R. Gene regulation by riboswitches[J]. Nat. Rev. Mol. Cell Biol., 2004, 5(6):451-463. |
| 55 | WINKLER W, NAHVI A, BREAKER R R. Thiamine derivatives bind messenger RNAs directly to regulate bacterial gene expression[J]. Nature, 2002, 419(6910):952-956. |
| 56 | ZHONG W, PU Y, TAN W, et al.. Identification and application of an aptamer targeting papillary thyroid carcinoma using tissue-SELEX[J]. Anal. Chem., 2019, 91(13):8289-8297. |
| 57 | LE A, KRYLOVA S M, KANOATOV M, et al.. Ideal-filter capillary electrophoresis (IFCE) facilitates the one-step selection of aptamers[J]. Angew. Chem., 2019, 131(9):2765-2769. |
| 58 | HUNG L Y, WANG C H, CHE Y J, et al.. Screening of aptamers specific to colorectal cancer cells and stem cells by utilizing On-chip Cell-SELEX[J]. Sci. Rep., 2015, 5(1):1-12. |
| 59 | LOU X, QIAN J, XIAO Y, et al.. Micromagnetic selection of aptamers in microfluidic channels[J]. Proc. Natl. Acad. Sci. USA, 2009, 106(9):2989-2994. |
| 60 | WEI Y Z, ZHANG W, LIU Z, et al.. Highly parallel single-molecule amplification approach based on agarose droplet polymerase chain reaction for efficient and cost-effective aptamer selection[J]. Anal. Chem., 2012, 84(1):350-355. |
| 61 | STEIN C A, CASTANOTTO D. FDA-approved oligonucleotide therapies in 2017[J]. J. Am. Soc. Gene Ther., 2017, 25(5):1069-1075. |
| 62 | GOLD L, AYERS D, BERTINO J, et al.. Aptamer-based multiplexed proteomic technology for biomarker discovery[J]. PLoS ONE, 2010, 5(12):1-17. |
| 63 | THIRUNAVUKARASU D, CHEN T, LIU Z, et al.. Selection of 2'-fluoro-modified aptamers with optimized properties[J]. J. Am. Chem. Soc., 2017, 139(8):2892-2895. |
| 64 | LIU H, MAI J, SHEN J, et al.. A novel DNA aptamer for dual targeting of polymorphonuclear myeloid-derived suppressor cells and tumor cells[J]. Theranostics, 2018, 8(1):31-44. |
| 65 | ROTHLISBERGER P, LEVI-ACOBAS F, SARAC I, et al.. On the enzymatic incorporation of an imidazole nucleotide into DNA[J]. Organ. Biomol. Chem., 2017, 15(20):4449-4455. |
| 66 | LIU M, YIN Q, CHANG Y, et al.. In vitro selection of circular DNA aptamers for biosensing applications[J]. Angew. Chem., 2019, 58(24):8013-8017. |
| [1] | 郎利新,窦晓莹,王欢,孔滢,白锦荣. 基于文献计量学的百合属植物研究态势分析[J]. 生物技术进展, 2020, 10(1): 96-107. |
| [2] | 卢垚,黄丹丹,王婷. 基于文献计量学的CGIAR对全球农业贡献研究[J]. 生物技术进展, 2019, 9(1): 89-97. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||
版权所有 © 2021《生物技术进展》编辑部